本文主要是介绍基于鸢尾花数据集的四种聚类算法(kmeans,层次聚类,DBSCAN,FCM)和学习向量量化对比,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
基于鸢尾花数据集的四种聚类算法(kmeans,层次聚类,DBSCAN,FCM)和学习向量量化对比
注:下面的代码可能需要做一点参数调整,才得到所有我的运行结果。
kmeans算法:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_score
iris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
#调用PCA
pca = PCA(n_components=2) # 降到2维
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵 (降维后的)
#X_dr#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)#plt.figure()
#plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
#plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
#plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
#plt.legend()
#plt.title('PCA of IRIS dataset')
#plt.show()print("===K-means聚类===")
from sklearn.cluster import KMeans # 引入KMeans模块estimator = KMeans(n_clusters=3).fit(X) # 构造聚类器
label_pred = estimator.labels_ # 获取聚类标签# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = []
# 遍历不同的n_clusters值
for n_clusters in range(2, 11): kmeans = KMeans(n_clusters=n_clusters) kmeans.fit(X) labels = kmeans.labels_ silhouette_avg = silhouette_score(X, kmeans.labels_) print(silhouette_avg)
# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, kmeans.labels_)print(calinski_haraba)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X, kmeans.labels_)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(range(2, 11), silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different n_clusters-kmeans')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(range(2, 11), Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title(' Calinski-Harabaszfor Different n_clusters-kmeans')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(range(2, 11), Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different n_clusters-kmeans')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()运行结果如下:
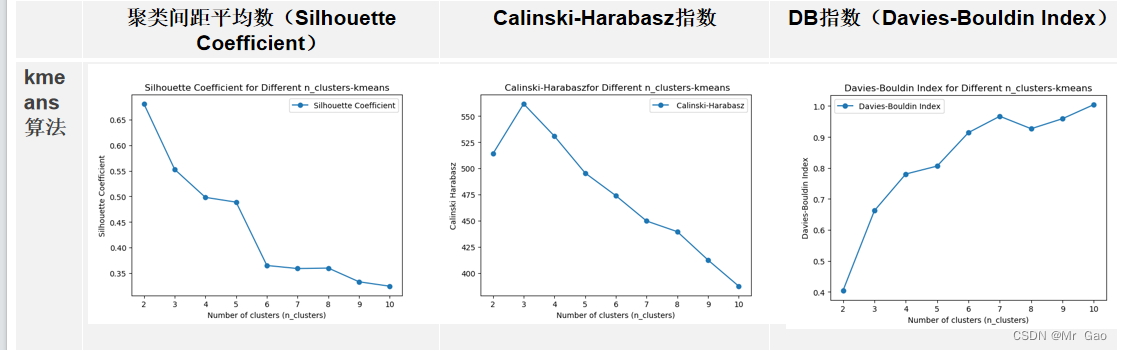
DBSCAN:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_scorefrom sklearn.cluster import DBSCAN # 引入DBSCAN模块iris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
##调用PCA
#pca = PCA(n_components=2) # 降到2维
#pca = pca.fit(X) #拟合模型
#X_dr = pca.transform(X) #获取新矩阵 (降维后的)
##X_dr#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)#plt.figure()
#plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
#plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
#plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
#plt.legend()
#plt.title('PCA of IRIS dataset')
#plt.show()print("===DBSCAN聚类===")
from sklearn.cluster import KMeans # 引入KMeans模块estimator = KMeans(n_clusters=3).fit(X) # 构造聚类器
label_pred = estimator.labels_ # 获取聚类标签# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = []
# 遍历不同的n_clusters值
for n_clusters in range(2, 11): dbscan = DBSCAN(eps=0.4, min_samples=n_clusters).fit(X) #导入DBSCAN模块进行训练,在一个邻域的半径内min_samples数的邻域eps被认为是一个簇。请记住,初始点包含在min_samples中。label_pred = dbscan.labels_ # labels为每个数据的簇标签,不在任何“高密度”集群中的“noisy”样本返回-1silhouette_avg = silhouette_score(X, dbscan.labels_) print(silhouette_avg)# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, dbscan.labels_)print(calinski_haraba)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X, dbscan.labels_)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(range(2, 11), silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different min_samples-DBSCAN-eps=0.4')
plt.xlabel('Number of min_samples (min_samples)')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(range(2, 11), Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title('Calinski-Harabasz for Different min_samples-DBSCAN-eps=0.4')
plt.xlabel('Number of min_samples (min_samples)')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(range(2, 11), Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different min_samples-DBSCAN-eps=0.4')
plt.xlabel('Number of min_samples (min_samples)')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = [] xindex= [0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9,1,1.1,1.2,1.4,1.6]
for s in xindex: print(s)dbscan = DBSCAN(eps=s, min_samples=3).fit(X) #导入DBSCAN模块进行训练,在一个邻域的半径内min_samples数的邻域eps被认为是一个簇。请记住,初始点包含在min_samples中。label_pred = dbscan.labels_ # labels为每个数据的簇标签,不在任何“高密度”集群中的“noisy”样本返回-1silhouette_avg = silhouette_score(X, dbscan.labels_) print(silhouette_avg)# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, dbscan.labels_)print(calinski_haraba)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X, dbscan.labels_)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(xindex, silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different min_samples-DBSCAN- min_samples=3')
plt.xlabel('eps')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(xindex, Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title('Calinski-Harabasz for Different min_samples-DBSCAN- min_samples=3')
plt.xlabel('eps')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(xindex, Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different min_samples-DBSCAN- min_samples=3')
plt.xlabel('eps')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()运行结果:
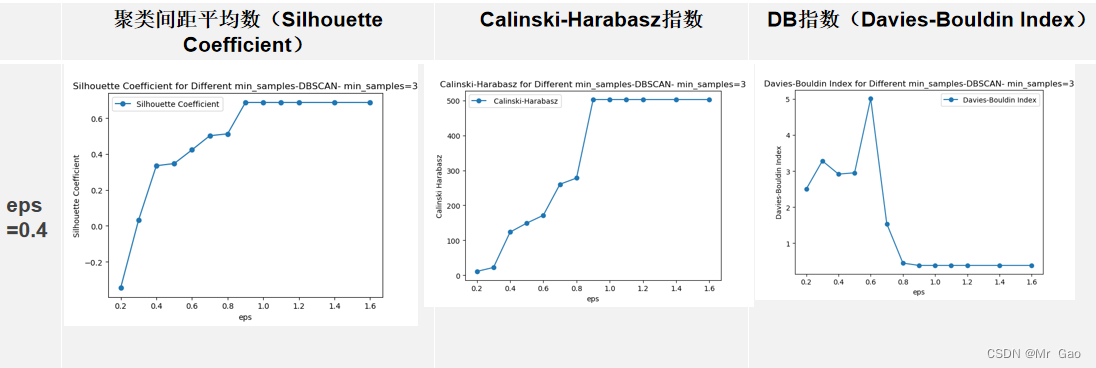
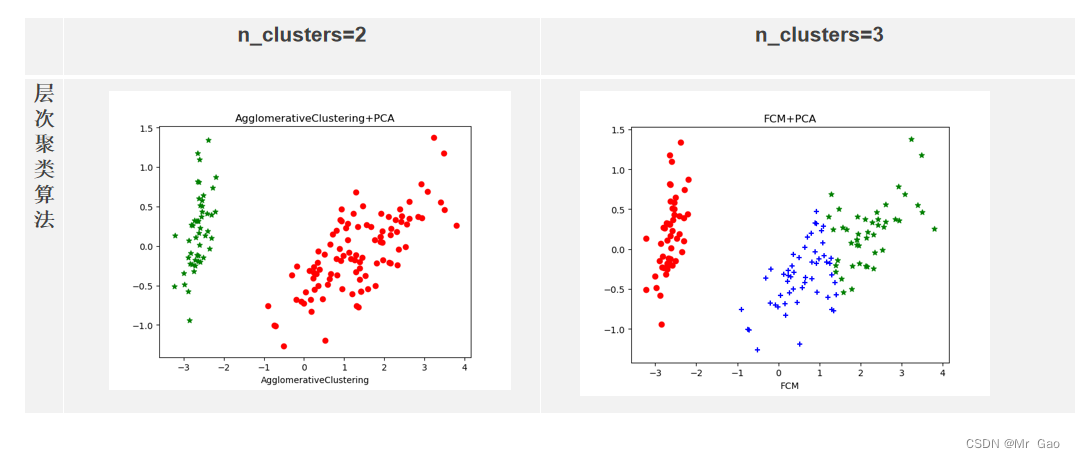
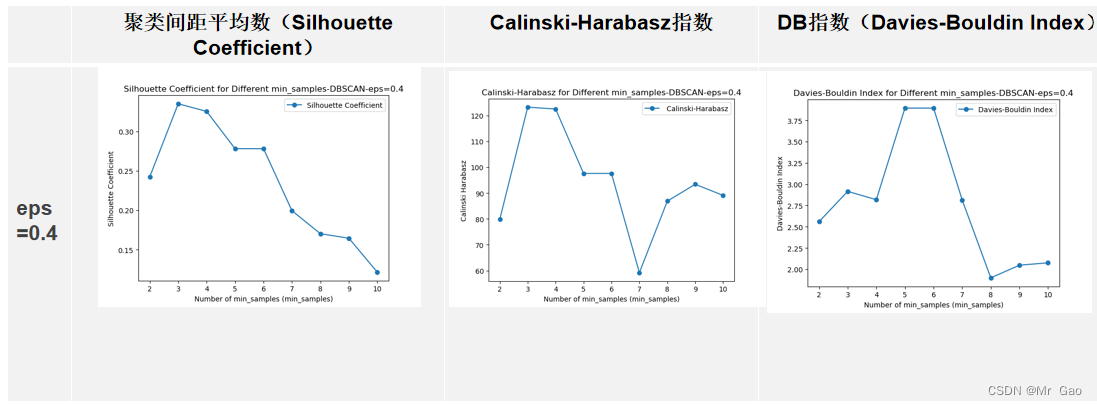 层次聚类:
层次聚类:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_scorefrom sklearn.cluster import AgglomerativeClustering
iris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
#调用PCA
pca = PCA(n_components=2) # 降到2维
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵 (降维后的)
#X_dr#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)#plt.figure()
#plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
#plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
#plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
#plt.legend()
#plt.title('PCA of IRIS dataset')
#plt.show()print("===K-means聚类===")
from sklearn.cluster import KMeans # 引入KMeans模块estimator = KMeans(n_clusters=3).fit(X) # 构造聚类器
label_pred = estimator.labels_ # 获取聚类标签# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = []
# 遍历不同的n_clusters值
for n_clusters in range(2, 11): agg = AgglomerativeClustering( n_clusters=n_clusters)agg.fit(X) labels = agg.labels_ silhouette_avg = silhouette_score(X, agg.labels_)
# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, agg.labels_)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X, agg.labels_)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(range(2, 11), silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different n_clusters-AgglomerativeClustering')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(range(2, 11), Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title(' Calinski-Harabaszfor Different n_clusters-AgglomerativeClustering')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(range(2, 11), Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different n_clusters-AgglomerativeClustering')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()运行结果:
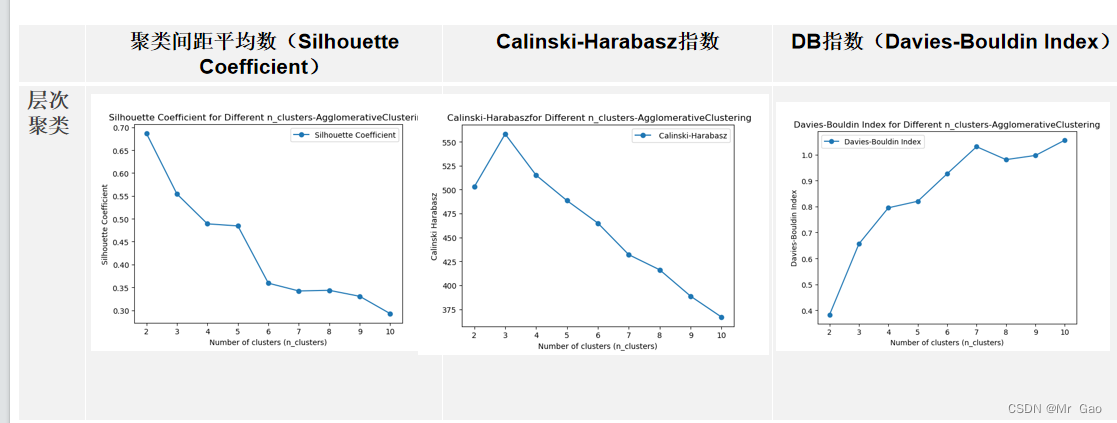
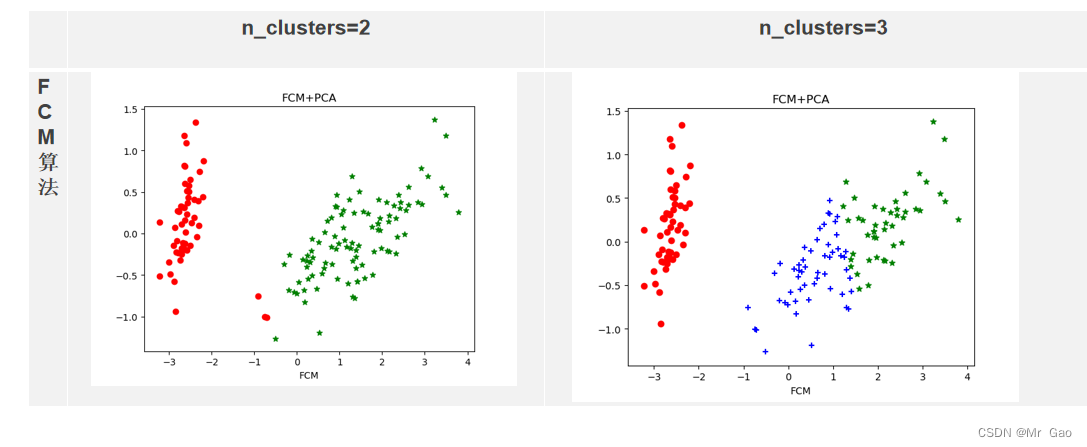
FCM算法:
代码:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_score
from sklearn.cluster import FeatureAgglomeration
from sklearn.cluster import AgglomerativeClustering
iris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
#调用PCA
pca = PCA(n_components=2) # 降到2维
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵 (降维后的)
#X_dr#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)#plt.figure()
#plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
#plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
#plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
#plt.legend()
#plt.title('PCA of IRIS dataset')
#plt.show()print("===K-means聚类===")
from sklearn.cluster import KMeans # 引入KMeans模块
def FCM(X, c_clusters=3, m=2, eps=10):membership_mat = np.random.random((len(X), c_clusters)) # 生成随机二维数组shape(150,3),随机初始化隶属矩阵# 这一步的操作是为了使Xi的隶属度总和为1membership_mat = np.divide(membership_mat, np.sum(membership_mat, axis=1)[:, np.newaxis])while True:working_membership_mat = membership_mat ** m # shape->(150,3)# 根据公式计算聚类中心点Centroids.shape->(3,4)Centroids = np.divide(np.dot(working_membership_mat.T, X), np.sum(working_membership_mat.T, axis=1)[:, np.newaxis])# 该矩阵保存所有实点到每个聚类中心的欧式距离n_c_distance_mat = np.zeros((len(X), c_clusters)) # shape->(150,3)for i, x in enumerate(X):for j, c in enumerate(Centroids):n_c_distance_mat[i][j] = np.linalg.norm(x-c, 2) # 计算l2范数(欧氏距离)new_membership_mat = np.zeros((len(X), c_clusters))# 根据公式计算模糊矩阵Ufor i, x in enumerate(X):for j, c in enumerate(Centroids):new_membership_mat[i][j] = 1. / np.sum((n_c_distance_mat[i][j] / n_c_distance_mat[i]) ** (2 / (m-1)))if np.sum(abs(new_membership_mat - membership_mat)) < eps:breakmembership_mat = new_membership_matreturn np.argmax(new_membership_mat, axis=1)# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = []
# 遍历不同的n_clusters值
for n_clusters in range(2, 11): print(n_clusters)fcm =FCM(X, c_clusters=n_clusters)print(len(fcm ))silhouette_avg = silhouette_score(X, fcm) print(silhouette_avg)
# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, fcm)print(calinski_haraba)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X,fcm)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(range(2, 11), silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different n_clusters-FCM')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(range(2, 11), Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title(' Calinski-Harabaszfor Different n_clusters-FCM')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(range(2, 11), Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different n_clusters-FCM')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()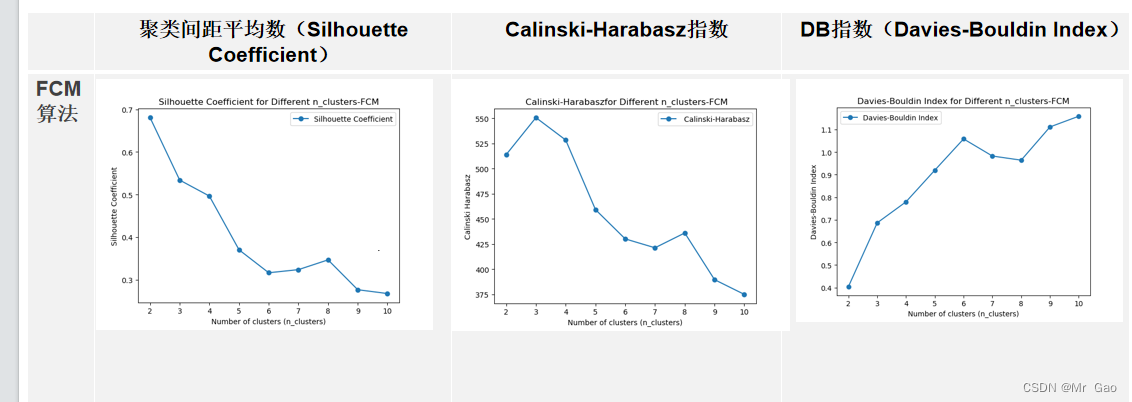
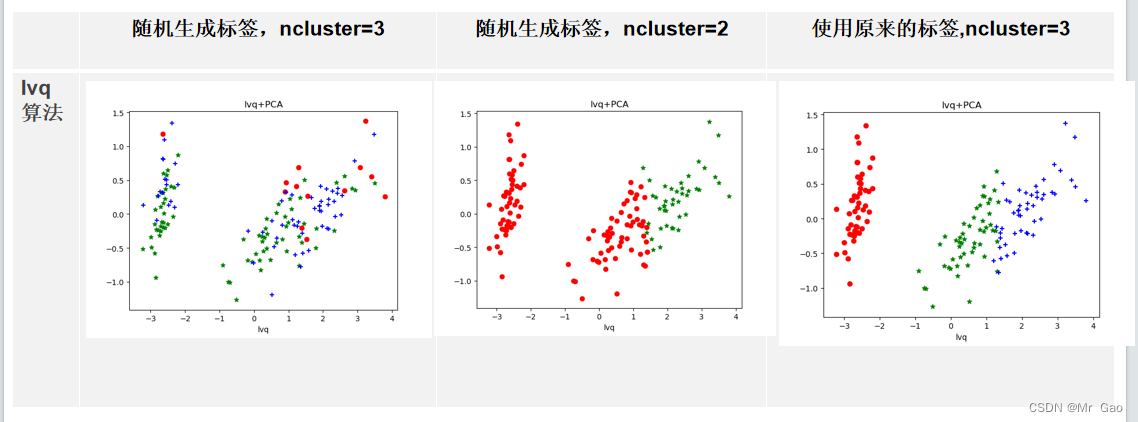
lvp算法:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
from sklearn.metrics import silhouette_score
from sklearn.metrics import calinski_harabasz_score
from sklearn.metrics import davies_bouldin_score
from sklearn.cluster import FeatureAgglomeration
from sklearn.cluster import AgglomerativeClustering# 使用LVQ进行聚类
from sklearn_lvq import GlvqModeliris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
#调用PCA
pca = PCA(n_components=2) # 降到2维
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵 (降维后的)
#X_dr#也可以fit_transform一步到位
#X_dr = PCA(2).fit_transform(X)#plt.figure()
#plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
#plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
#plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
#plt.legend()
#plt.title('PCA of IRIS dataset')
#plt.show()
def FCM(X, c_clusters=3, m=2, eps=10):membership_mat = np.random.random((len(X), c_clusters)) # 生成随机二维数组shape(150,3),随机初始化隶属矩阵# 这一步的操作是为了使Xi的隶属度总和为1membership_mat = np.divide(membership_mat, np.sum(membership_mat, axis=1)[:, np.newaxis])while True:working_membership_mat = membership_mat ** m # shape->(150,3)# 根据公式计算聚类中心点Centroids.shape->(3,4)Centroids = np.divide(np.dot(working_membership_mat.T, X), np.sum(working_membership_mat.T, axis=1)[:, np.newaxis])# 该矩阵保存所有实点到每个聚类中心的欧式距离n_c_distance_mat = np.zeros((len(X), c_clusters)) # shape->(150,3)for i, x in enumerate(X):for j, c in enumerate(Centroids):n_c_distance_mat[i][j] = np.linalg.norm(x-c, 2) # 计算l2范数(欧氏距离)new_membership_mat = np.zeros((len(X), c_clusters))# 根据公式计算模糊矩阵Ufor i, x in enumerate(X):for j, c in enumerate(Centroids):new_membership_mat[i][j] = 1. / np.sum((n_c_distance_mat[i][j] / n_c_distance_mat[i]) ** (2 / (m-1)))if np.sum(abs(new_membership_mat - membership_mat)) < eps:breakmembership_mat = new_membership_matreturn np.argmax(new_membership_mat, axis=1)# 评估指标列表
silhouette_avg_scores = [] Calinski_Harabasz_scores = []
Davies_Bouldin_scores = []
from sklearn.datasets import make_blobs# 遍历不同的n_clusters值
for n_clusters in range(2, 11): print(n_clusters)zX, y_true = make_blobs(n_samples=150, centers=n_clusters, cluster_std=0.6, random_state=0)lvq = GlvqModel()lvq.fit(X, y_true)# 可视化聚类结果fcm = lvq.predict(X)print(len(fcm ))silhouette_avg = silhouette_score(X, fcm) print(silhouette_avg)
# 2. Calinski-Harabasz指数calinski_haraba=calinski_harabasz_score(X, fcm)print(calinski_haraba)# 3. DB指数(Davies-Bouldin Index)davies_bouldin=davies_bouldin_score(X,fcm)Davies_Bouldin_scores.append(davies_bouldin)Calinski_Harabasz_scores.append(calinski_haraba)silhouette_avg_scores.append(silhouette_avg) # 绘制图形
plt.plot(range(2, 11), silhouette_avg_scores, marker='o', label='Silhouette Coefficient') plt.title('Silhouette Coefficient for Different n_clusters--lvp')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Silhouette Coefficient')
plt.legend()
plt.show()plt.plot(range(2, 11), Calinski_Harabasz_scores, marker='o', label=' Calinski-Harabasz')
plt.title(' Calinski-Harabaszfor Different n_clusters--lvp')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Calinski Harabasz')
plt.legend()
plt.show()plt.plot(range(2, 11), Davies_Bouldin_scores, marker='o', label='Davies-Bouldin Index')
plt.title('Davies-Bouldin Index for Different n_clusters-lvp')
plt.xlabel('Number of clusters (n_clusters)')
plt.ylabel('Davies-Bouldin Index')
plt.legend()
plt.show()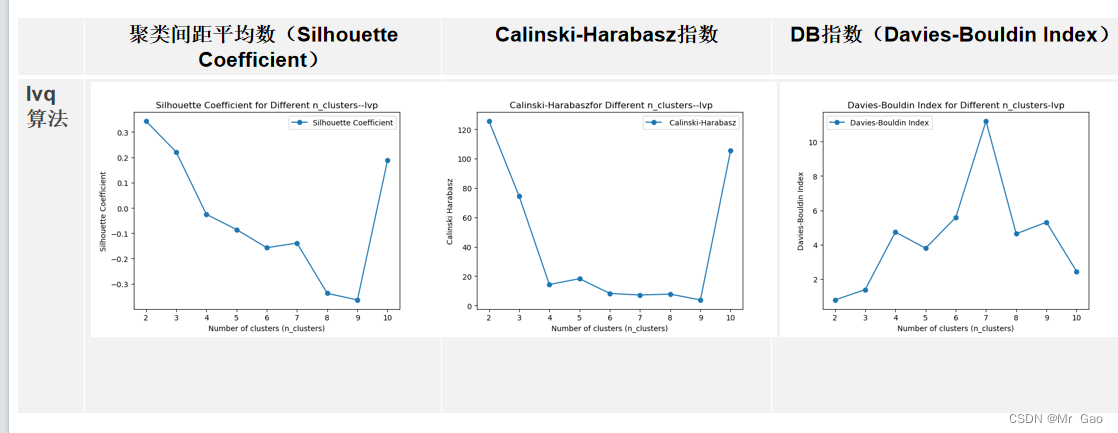
最后我们还做了一个所有算法最优参数汇总的代码:
import matplotlib.pyplot as plt # 导入matplotlib的库
import numpy as np # 导入numpy的包
from sklearn import datasets #导入数据集
from sklearn.decomposition import PCA # PCA主成分分析类
iris = datasets.load_iris() #加载iris数据集
X = iris.data #加载特征数据
# Y = iris.target #加载标签数据
#绘制数据分布图
y = iris.target
X = iris.data
#X.shape
#调用PCA
pca = PCA(n_components=2) # 降到2维
pca = pca.fit(X) #拟合模型
X_dr = pca.transform(X) #获取新矩阵 (降维后的)
#X_dr#也可以fit_transform一步到位
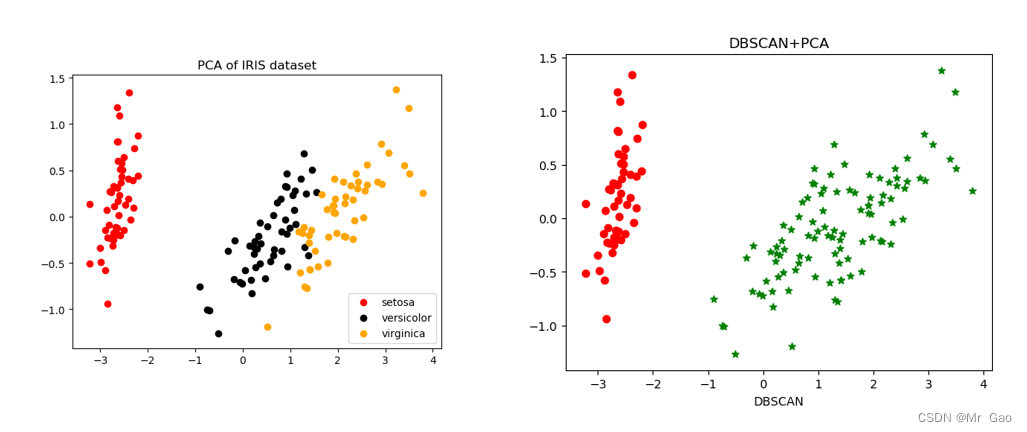
X_dr = PCA(2).fit_transform(X)plt.figure()
plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
plt.legend()
plt.title('PCA of IRIS dataset')
plt.show()print("===K-means聚类===")
from sklearn.cluster import KMeans # 引入KMeans模块
estimator = KMeans(n_clusters=3).fit(X) # 构造聚类器
label_pred = estimator.labels_ # 获取聚类标签
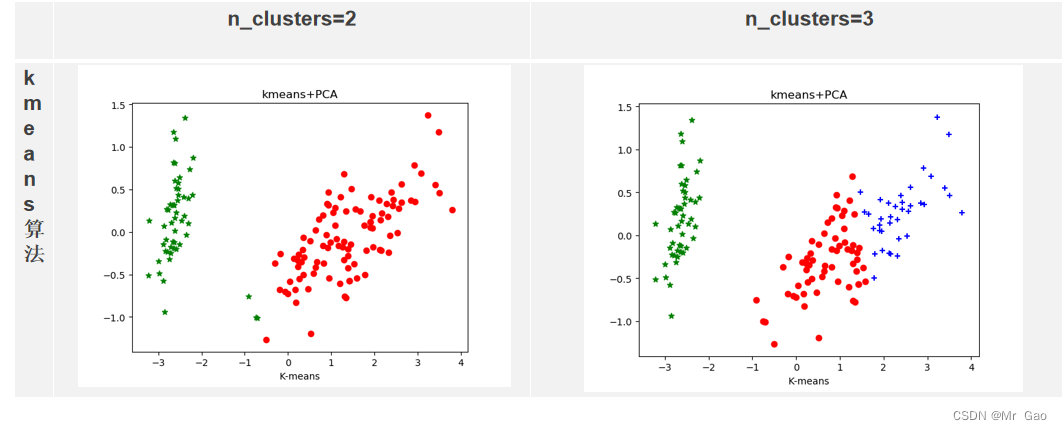
#绘制k-means结果
x0 = X_dr[label_pred == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X_dr[label_pred == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X_dr[label_pred == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('K-means')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("kmeans+PCA")
plt.show()x0 = X[label_pred == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X[label_pred == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X[label_pred == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画laplt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('K-means')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("kmeans-features[0:2]")
plt.show()#密度聚类之DBSCAN算法
print("===DBSCAN聚类===")
from sklearn.cluster import DBSCAN # 引入DBSCAN模块
dbscan = DBSCAN(eps=1.0, min_samples=3).fit(X) #导入DBSCAN模块进行训练,在一个邻域的半径内min_samples数的邻域eps被认为是一个簇。请记住,初始点包含在min_samples中。
label_pred = dbscan.labels_ # labels为每个数据的簇标签,不在任何“高密度”集群中的“noisy”样本返回-1x0 = X[label_pred == 0] # 获取聚类标签等于0的话,则赋值给x0
x1 = X[label_pred == 1] # 获取聚类标签等于1的话,则赋值给x1
x2 = X[label_pred == 2] # 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label0') # 画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label1') # 画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label2') # 画label 2的散点图
plt.xlabel('DBSCAN')# 设置X轴的标签为DBSCAN
plt.legend(loc=2)# 设置图标在左上角
plt.title("DBSCAN-features[0:2]")
plt.show()x0 = X_dr[label_pred == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X_dr[label_pred == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X_dr[label_pred == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('DBSCAN')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("DBSCAN+PCA")
plt.show()
from sklearn_lvq import GlvqModelimport numpy as np
from matplotlib import pyplot as plt
from scipy.cluster.hierarchy import dendrogram
from scipy.cluster.hierarchy import linkage, dendrogram
def getLinkageMat(model):children = model.children_cs = np.zeros(len(children))N = len(model.labels_)for i,child in enumerate(children):count = 0for idx in child:count += 1 if idx < N else cs[idx - N]cs[i] = countreturn np.column_stack([children, model.distances_, cs])from sklearn.cluster import AgglomerativeClustering
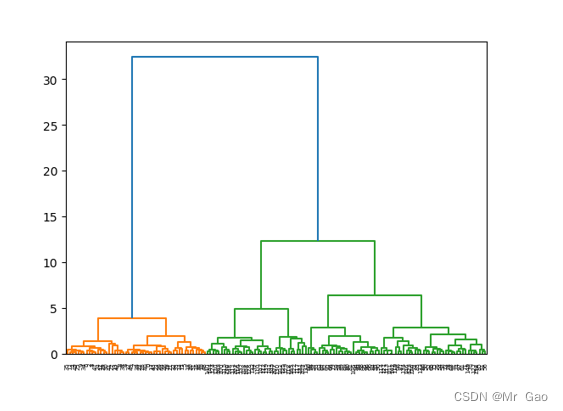
from sklearn.datasets import make_blobsmodel = AgglomerativeClustering( n_clusters=3)model = model.fit(X)label_pred = model.labels_ # labels为每个数据的簇标签,不在任何“高密度”集群中的“noisy”样本返回-1Z = linkage(X, method='ward', metric='euclidean')
p = dendrogram(Z, 0)
plt.show()x0 = X_dr[label_pred == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X_dr[label_pred == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X_dr[label_pred == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('AgglomerativeClustering')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("AgglomerativeClustering+PCA")
plt.show()def FCM(X, c_clusters=3, m=2, eps=10):membership_mat = np.random.random((len(X), c_clusters)) # 生成随机二维数组shape(150,3),随机初始化隶属矩阵# 这一步的操作是为了使Xi的隶属度总和为1membership_mat = np.divide(membership_mat, np.sum(membership_mat, axis=1)[:, np.newaxis])while True:working_membership_mat = membership_mat ** m # shape->(150,3)# 根据公式计算聚类中心点Centroids.shape->(3,4)Centroids = np.divide(np.dot(working_membership_mat.T, X), np.sum(working_membership_mat.T, axis=1)[:, np.newaxis])# 该矩阵保存所有实点到每个聚类中心的欧式距离n_c_distance_mat = np.zeros((len(X), c_clusters)) # shape->(150,3)for i, x in enumerate(X):for j, c in enumerate(Centroids):n_c_distance_mat[i][j] = np.linalg.norm(x-c, 2) # 计算l2范数(欧氏距离)new_membership_mat = np.zeros((len(X), c_clusters))# 根据公式计算模糊矩阵Ufor i, x in enumerate(X):for j, c in enumerate(Centroids):new_membership_mat[i][j] = 1. / np.sum((n_c_distance_mat[i][j] / n_c_distance_mat[i]) ** (2 / (m-1)))if np.sum(abs(new_membership_mat - membership_mat)) < eps:breakmembership_mat = new_membership_matreturn np.argmax(new_membership_mat, axis=1)fcm =FCM(X, c_clusters=3)x0 = X_dr[fcm == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X_dr[fcm == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X_dr[fcm == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('FCM')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("FCM+PCA")
plt.show()zX, y_true = make_blobs(n_samples=150, centers=2, cluster_std=0.6, random_state=0)
lvq = GlvqModel()
lvq.fit(X, y)# 可视化聚类结果
lvqp = lvq.predict(X)x0 = X_dr[lvqp == 0]# 获取聚类标签等于0的话,则赋值给x0
x1 = X_dr[lvqp == 1]# 获取聚类标签等于1的话,则赋值给x1
x2 = X_dr[lvqp == 2]# 获取聚类标签等于2的话,则赋值给x2
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label 0')#画label 0的散点图
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label 1')#画label 1的散点图
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label 2')#画label 2的散点图
plt.xlabel('lvq')# 设置X轴的标签为K-means
# plt.legend(loc=2)# 设置图标在左上角
plt.title("lvq+PCA")
plt.show()运行结果:
这篇关于基于鸢尾花数据集的四种聚类算法(kmeans,层次聚类,DBSCAN,FCM)和学习向量量化对比的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




