本文主要是介绍NASA数据集——北美地区永久冻土影响的冻原和北方生态系统内发生的土壤呼吸作用产生的二氧化碳(CO2)排放量(300 米的空间分辨率),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Soil Respiration Maps for the ABoVE Domain, 2016-2017
简介
文件修订日期:2022-04-20
数据集版本: 1
摘要
该数据集以 300 米的空间分辨率提供了 2016-08-18 至 2018-09-12 期间阿拉斯加和加拿大西北部受永久冻土影响的冻原和北方生态系统内发生的土壤呼吸作用产生的二氧化碳(CO2)排放量的网格估算值。估算结果包括月平均二氧化碳通量(gCO2 C m-2 d-1)、按季节(秋季、冬季、春季、夏季)划分的日平均二氧化碳通量和误差估算值、二氧化碳年吸收偏移量(即植被总初级生产力)估算值、植被总初级生产力年度预算(GPP;gCO2 C m-2 yr-1)以及每个 300 米网格单元内开放(非植被)水域的比例。地下呼吸源(即根和微生物)也包括在内。网格化土壤二氧化碳估算值是利用季节性随机森林模型、遥感信息以及来自土壤呼吸站和涡度协方差塔的原位土壤二氧化碳通量新汇编获得的。通量塔数据与每个土壤呼吸站强制扩散(FD)室记录的每日间隙通量观测数据一起提供。数据覆盖 NASA ABoVE 域。
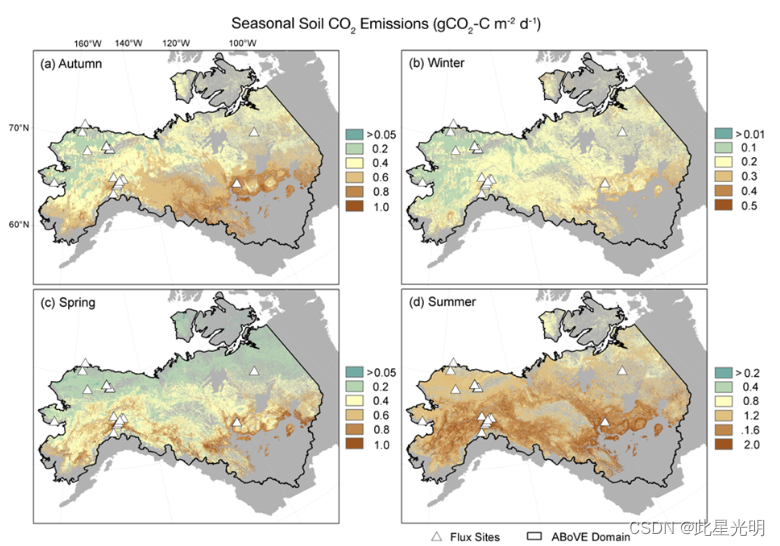
该数据集有 28 个数据文件;25 个数据文件为 GeoTIFF(*.tif)格式,3 个文件为逗号分隔值格式(.csv)。下图为空间分辨率为 300 米的秋季(9 月-10 月)、冬季(11 月-3 月)、春季(4 月-5 月)和夏季(6 月-8 月)的季节性平均土壤呼吸排放量(gCO2 C m-2 d-1)。资料来源:Watts et al:Watts 等人(2021 年)
项目北极-北方脆弱性实验
北极-北方脆弱性实验(ABoVE)是美国国家航空航天局(NASA)陆地生态计划的一项实地活动,重点是阿拉斯加和加拿大西部。ABoVE 的研究将基于实地的过程级研究与机载和卫星传感器获得的地理空间数据产品联系起来,为提高分析和建模能力奠定了基础,这些能力是了解和预测北极和北方地区生态系统对气候变化的反应以及气候变化对社会的影响所必需的。我们由 ABoVE 赞助的研究重点是土壤二氧化碳的时空模式和量级,而在迅速变暖的泛北极生态系统中,这些模式和量级还没有得到很好的量化。
数据特征
空间覆盖范围:阿拉斯加和加拿大受永久冻土影响的冻原和北方地貌(ABoVE 核心域);不包括荒地和水体
ABoVE 参考地点
域:ABoVE 核心域
网格单元(240 米 "A "单元):Ah000v000、Ah000v00、Ah001v002、Ah002v002、Ah003v002、Ah001v000、Ah002v000、Ah003v000、
Ah000v001、Ah001v001、Ah002v001、Ah003v001
空间分辨率:300 米网格单元,站点数据的点分辨率
时间覆盖范围:2016-08-18 至 2018-09-12
时间分辨率:日、月
研究区域:纬度和经度均以十进制度表示。
| Site | Westernmost Longitude | Easternmost Longitude | Northernmost Latitude | Southernmost Latitude |
|---|---|---|---|---|
| Alaska and Canada | -169.5052 | -98.7366 | 76.6874 | 55.8072 |
数据文件信息
该数据集中包含 25 个 GeoTIFF (*.tif) 格式的数据文件和 3 个逗号分隔值 (*.csv) 格式的文件。
| File Name | Units | Description |
|---|---|---|
| soil_resp_flux_YYYY-XX.tif | g m-2 d-1 | Twelve files. Daily average emissions from soil respiration (gCO2 C m-2 d-1) over monthly time steps where YYYY is 2016 or 2017 and XX for the 2016 files is 09–12, and for the 2017 files is 01–08. Missing values are represented by 0. |
| soil_resp_flux_AAA_YYYY.tif | g m-2 d-1 | Four files. Average estimates (gCO2 C m-2 d-1) for sampling periods, where AAA is the season and YYYY is the year. Missing values are represented by 0. |
| soil_resp_flux_est_error_AAA_YYYY.tif | g m-2 d-1 | Four files. Average flux estimate error (gCO2 C m-2 d-1) for sampling periods, where AAA is the season and YYYY is the year. Missing values are represented by -3. |
| soil_resp_budget_annual_2016-2017.tif | g m-2 | Annual soil respiration budgets (gCO2 C m-2) for September 2016–August 2017. Missing values are represented by 0. |
| soil_resp_budget_annual_percent_offset_GPP_2016-2017.tif | percent | Annual percentage offset of growing season GPP by soil respiration emissions for September 2016–August 2017. An offset of 100% indicates that soil respiration equaled or exceeded annual GPP. Missing values are represented by 0. |
| GPP_budget_annual_2016-2017.tif | g m-2 | Annual GPP budgets (gCO2 C m-2) averaged for the 2016–2017 growing seasons. A composite of SMAP, GOSIF, and MODIS GPP products. Missing values are represented by 0. |
| ecosystem_resp_budget_annual_percent_offset_GPP_2016-2017.tif | percent | Annual percentage offset of growing season GPP by ecosystem (soil + aboveground) respiration forSeptember 2016–August 2017. An offset of 100% indicates that ecosystem respiration equaled or exceeded annual GPP. Missing values are represented by 0. |
| fractional_water.tif | 1 | Provides 300 m grid fractional water (FW) information for the study domain, derived from Wang et al. (2019). FW values of 1 indicate that the 300 m pixel contains 100% surface open water coverage. Missing values are represented by 0. |
| soil_resp_station_descriptions.csv | Provides descriptive data of site respiration stations. See Table 2 for variable descriptions. Missing values are represented by -9999. | |
| site_monthly_soil_respiration_flux.csv | Table of average monthly soil respiration fluxes for the ABoVE domain in gCO2 C m-2 d-1, used to develop the Random Forest models. These include flux data from Soil Respiration Stations and eddy covariance sites for 2016–2017 (Watts et al., 2021). The Soil Respiration Station record also includes more recent data for 2018. See Table 3 for variable descriptions. Missing values are represented by -9999. | |
| soil_resp_station_daily_soil_respiration_flux_gap_filled_2016-2018.csv | Table of daily gap filled flux observations for each Alaska Soil Respiration station forced diffusion (FD) chamber record (Watts et al., 2021). The record includes all available data from 2016–2018. See Table 4 for variable descriptions. Missing values are represented by -9999. |
数据细节
缩放因子:对于文件 fractional_water.tif(水遮罩),缩放因子为 100。
CRS:圆柱等面积,WGS 84 基准,EPSG:6933。
缺失数据值:对于名为 soil_resp_flux_est_error_AAA_YYYY.tif 的文件,缺失值以 -3 表示;对于其他 GeoTIFF 文件,缺失值以 0 表示;对于 CSV 文件,缺失值以 -9999 表示。
文件 soil_resp_station_descriptions.csv 中的变量名称和说明。
| Variable | Units | Description |
|---|---|---|
| site_code | Abbreviated site station name | |
| site_name | Site station name | |
| latitude | Decimal degrees | Latitude of site |
| longitude | Decimal degrees | Longitude of site |
| description | Site description (vegetation type, burned information) |
site_monthly_soil_respiration_flux.csv 文件中的变量名称和说明。
| Variable | Units | Description |
|---|---|---|
| site | Abbreviated site station name | |
| system | System used to collect data: EC (eddy covariance towers), SRS (Soil Respiration Stations), or Eosense FD (forced diffusion chamber) | |
| latitude | Decimal degrees | Latitude of site |
| longitude | Decimal degrees | Longitude of site |
| month | Month of measurement | |
| flux | g m-2 d-1 | Monthly average flux observations (gCO2 C m-2 d-1) from Soil Respiration Stations and eddy covariance towers |
文件 soil_resp_station_daily_soil_respiration_flux_gap_filled_2016-2018.csv 的变量名称和说明。
| Variable | Units | Description |
|---|---|---|
| date | Abbreviated site station name | |
| XXX_FDZZ (site code names_chamber record) | g m-2 d-1 | Gap-filled flux data (gCO2 C m-2 d-1) for 31 respiration sites, in columns B–AF, for each soil respiration station forced diffusion (FD_ZZ) chamber record. For site code names, refer to Table 2. |
应用与推导
土壤排放和预算图为了解域内土壤呼吸的区域模式提供了新的视角。由此得出的呼吸预算可用于与基于过程的区域模型和地球系统模型的估计值进行基线比较。
质量评估
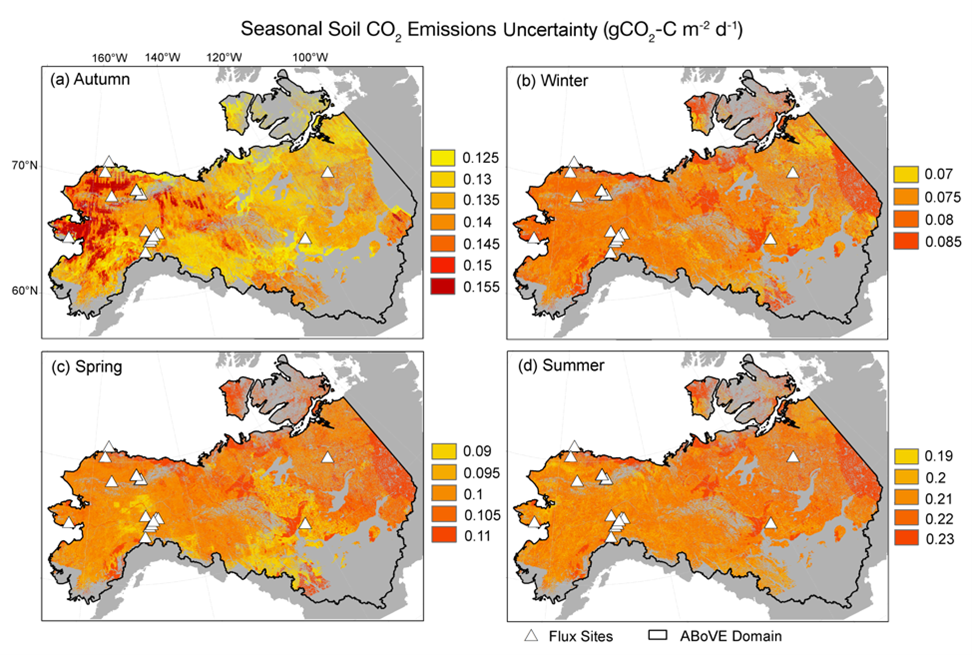
土壤呼吸排放量来自使用 ABoVE 域新二氧化碳通量数据库(Watts 等,2021 年)训练的模型。通量数据库包括来自 10 个土壤呼吸站(Minions 等,2019 年)、1 个 Eosense eosFD 站和 15 个涡度协方差塔的观测数据。从 2016 年 9 月到 2017 年 8 月,共有 268 个月平均通量(gCO2 C m-2 d-1)估算值被用于开发区域土壤呼吸模型。根据内部和外部精度评估,使用均方根误差(RMSE)、判定系数(R2)和平均绝对误差(MAE)对随机森林模型误差的不确定性进行了测量(表 2)。每个季节性随机森林模型的区域不确定性图(图 3)是用正交法得到的:sqrt(全局 RMSE2 + (像素级不确定性-全局 RMSE)2)。像素级不确定性是通过对随机森林输入观测数据(去掉 20%)进行自举得到的,以估算出估计平均值周围的偏差(不确定性)。
季节性随机森林模型的精度评估。RMSE1和MAE1(gCO2 C m-2 d-1)来自内部样本外精度评估;RMSE2和MAE2基于外部精度评估,使用1000个引导样本,其中80%的数据用于模型训练,20%的数据不用于验证。
| Season | R2 | RMSE1 | MAE1 | RMSE2 | MAE2 | |||||
|---|---|---|---|---|---|---|---|---|---|---|
| Summer | 0.68 | 0.16 | 0.05 | 0.35 | 0.31 | |||||
| Autumn | 0.57 | 0.12 | 0.09 | 0.24 | 0.20 | |||||
| Winter | 0.65 | 0.06 | 0.04 | 0.10 | 0.08 | |||||
| Spring | 0.76 | 0.08 | 0.11 | 0.25 | 0.19 | |||||
数据获取、材料和方法
土壤呼吸排放量是利用 ABoVE 域的新二氧化碳通量数据库(Watts 等人,2021 年)得出的,该数据库包括来自 10 个土壤呼吸站(Minions 等人,2019 年)、1 个 Eosense eosFD 站和 15 个涡度协方差塔的观测数据。通量数据库总共提供了 268 个月平均通量观测值(gCO2 C m-2 d-1),时间跨度为 2016 年 9 月至 2017 年 8 月。其中 14 个站点位于苔原生态系统;12 个站点位于北方生物群落;3 个站点位于焚烧生态系统。
代码
!pip install leafmap
!pip install pandas
!pip install folium
!pip install matplotlib
!pip install mapclassifyimport pandas as pd
import leafmapurl = "https://github.com/opengeos/NASA-Earth-Data/raw/main/nasa_earth_data.tsv"
df = pd.read_csv(url, sep="\t")
dfleafmap.nasa_data_login()results, gdf = leafmap.nasa_data_search(short_name="ABoVE_Soil_Respiration_Maps_1935",cloud_hosted=True,bounding_box=(-169.51, 55.81, -98.74, 76.69),temporal=("2016-08-18", "2018-09-12"),count=-1, # use -1 to return all datasetsreturn_gdf=True,
)gdf.explore()#leafmap.nasa_data_download(results[:5], out_dir="data")引用
Watts, J.D., S. Natali, and C. Minions. 2022. Soil Respiration Maps for the ABoVE Domain, 2016-2017. ORNL DAAC, Oak Ridge, Tennessee, USA. Soil Respiration Maps for the ABoVE Domain, 2016-2017, https://doi.org/10.3334/ORNLDAAC/1935
网址推荐
0代码在线构建地图应用
https://sso.mapmost.com/#/login?source_inviter=nClSZANO
机器学习
https://www.cbedai.net/xg
这篇关于NASA数据集——北美地区永久冻土影响的冻原和北方生态系统内发生的土壤呼吸作用产生的二氧化碳(CO2)排放量(300 米的空间分辨率)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




