本文主要是介绍分子对接教程 | (7) AutoDock对接中易错问题,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!

TCGA | GEO | 文献阅读 | 数据库 | 理论知识
R语言 | Bioconductor | 服务器与Linux

接前文:
分子对接教程 | (1) 软件安装准备
分子对接教程 | (2) 选择合适的蛋白受体
分子对接教程 | (3) 配体分子文件格式转换
分子对接教程 | (4) 蛋白受体文件的预处理
分子对接教程 | (5) 配体小分子的预处理
分子对接教程 | (6) AutoDock对接操作与对接结果解读
有些同学可能第一次做对接,由于蛋白或者的受体不同,中途可能会产生一些错误,导致后面无法进行,一直停留在原地,这里,我简单介绍一下,可能的出现的错误,但不一定覆盖到你所遇到的问题。
首先就是准备受体的时候,加氢的问题,其实在前文已经有过介绍,在选择一个分子作为配体或受体之前,必须把所有的氢都加到这个分子上,而且是全氢。如果你操作是用我选择的蛋白或者受体,前面可能你没有发现一个问题,那就小分子配体,我们下载的sdf文件和mol2文件,其中的sdf文件在PubChem数据库下载的,在3D Conformer处左侧有一些选项勾选,比如是否显示氢键,我当时是勾选的。
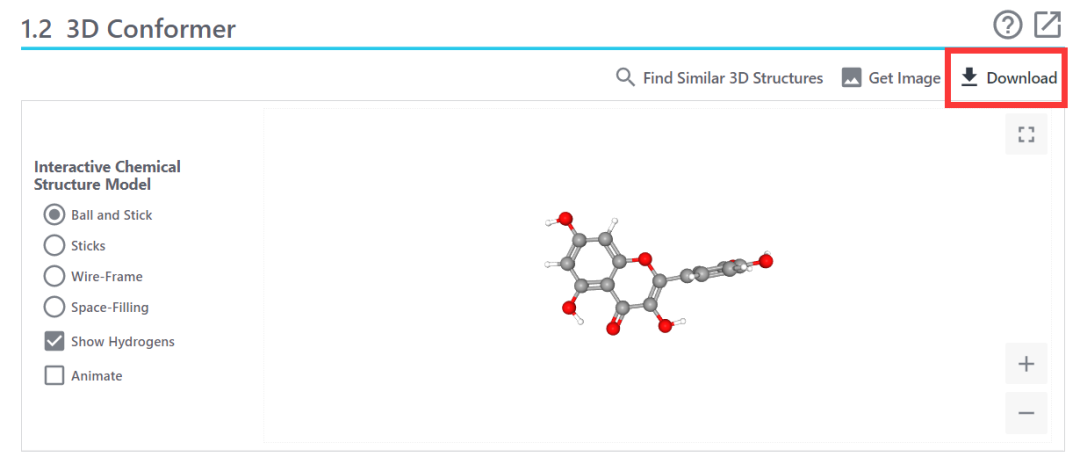
如果我们的小分子已经带氢,再加氢是没有什么变化的。

当然,这一步没有什么影响。
还有就是我们是否计算Gasteiger电荷,这在前面也有所提到,对于小分子配体,我们是不自己去计算的,除非出错,这在前面有解释,但对于蛋白配体呢??是否也需要计算Gasteiger电荷??这里是不需要的,我们在去水加氢后选为受体,软件是自动添加,所以,对于蛋白质来说,无论是否报错,均不能计算Gasteiger电荷这一步操作,这会改变真正的对接结果。
但是对于蛋白受体来说,一般不会出错的,要出错的情况最可能的原因就是你蛋白受体含有特殊原子,而这些离子在AutoDockTools的离子参数配置文件中没有,所以会报错,这种怎么解决??
我们首先先看一下原子参数的配置文件,如果你安装软件的时候没有改变安装路径的话,应该在C:\Program Files (x86)\MGLTools-1.5.6\MGLToolsPckgs\AutoDockTools 这个路径下。
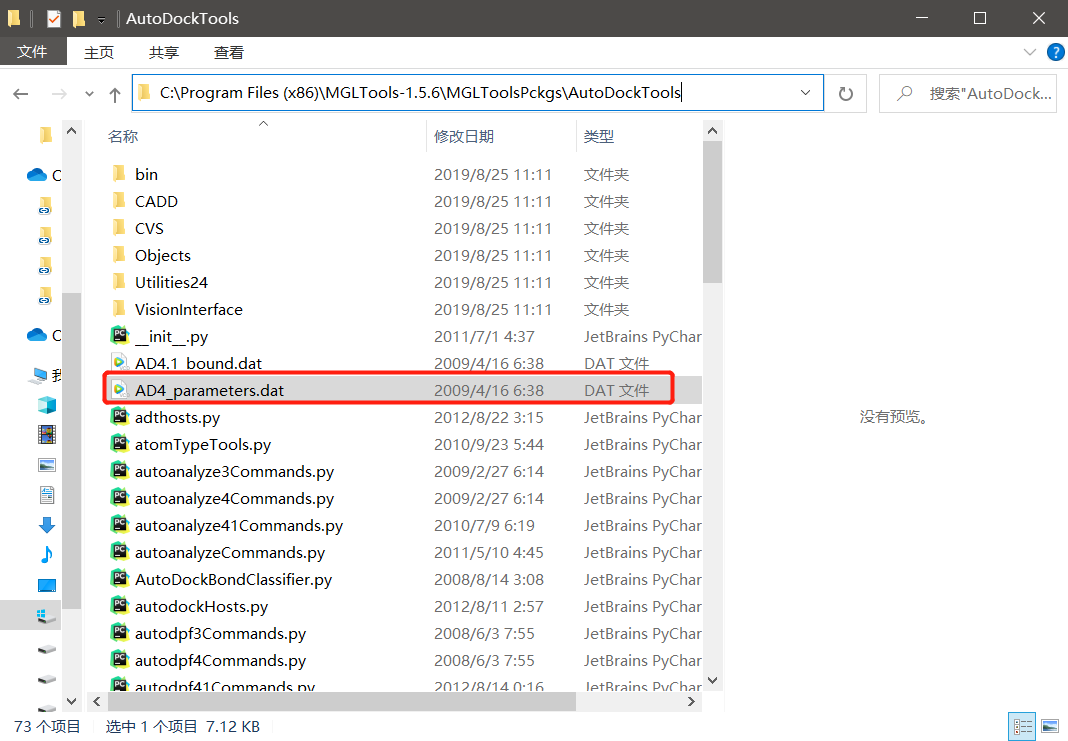
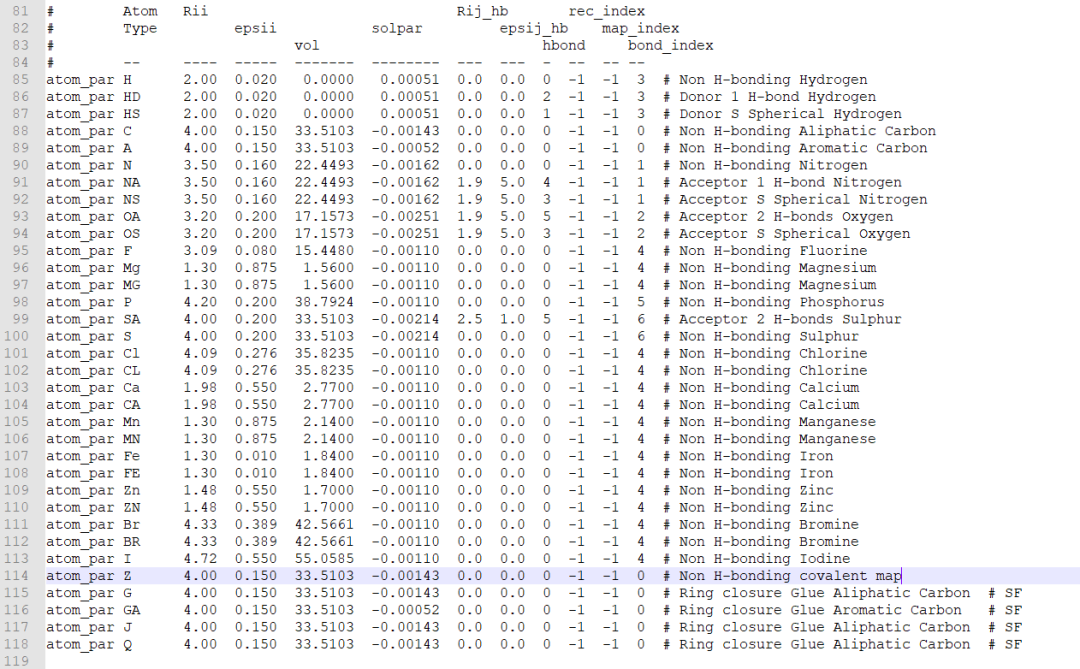
AD4_parameters.dat这个文件就是原子参数配置文件,不懂的话先不要更改它,你复制一份出来,用文本软件打开,你会找到如下的内容。这些就是已有的离子参数,你看一下里面有没有你的金属离子,没有的话报错的原因就是这个。
如 3PAU这个蛋白,含有铜离子,而铜离子在我们默认的参数里面是没有的。
我们下载这个蛋白用ADT打开。去水加氢。
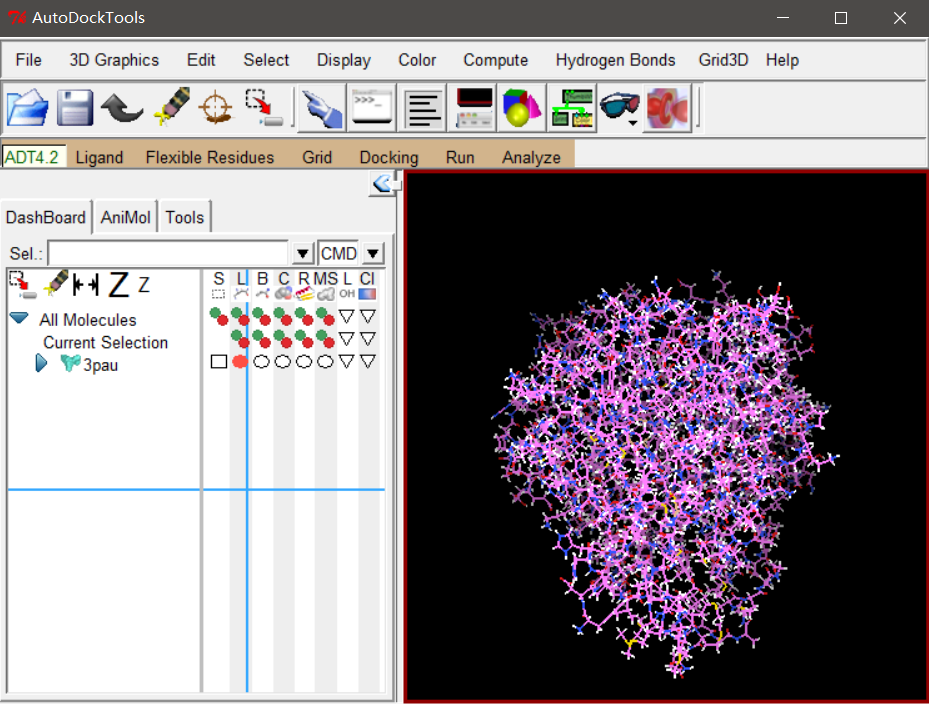
我们接着讲蛋白设置为受体,准备输出为pdbqt文件时,认真的会发现,和前面有所不同,会提示一些原子有0个电荷,这种提示在前面是没有出现的。
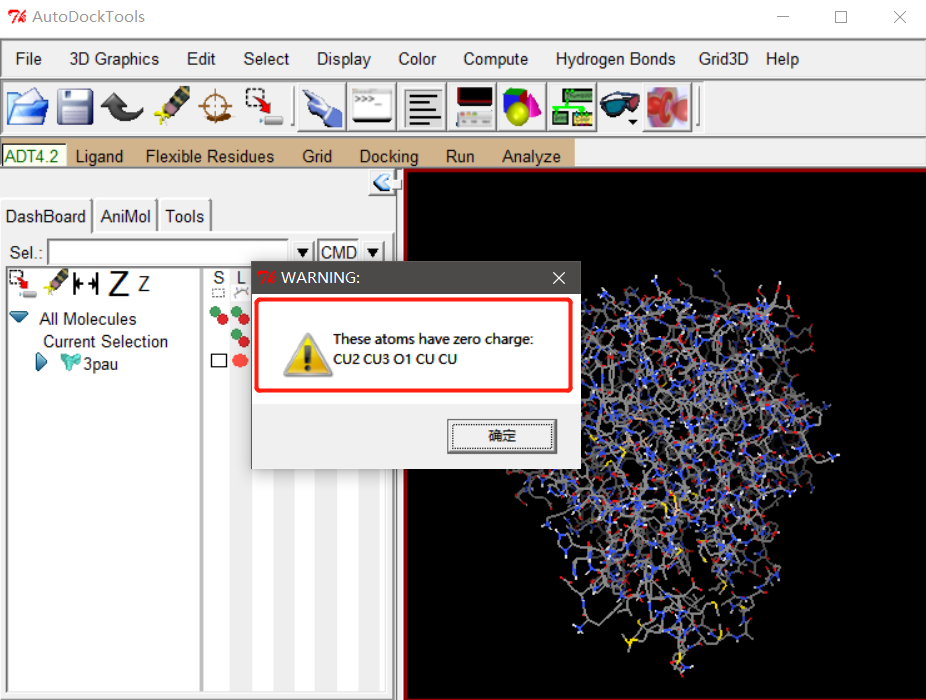
我们按照前面的操作流程,导入受体分子pdbqt文件时,还是会有类似的提示。
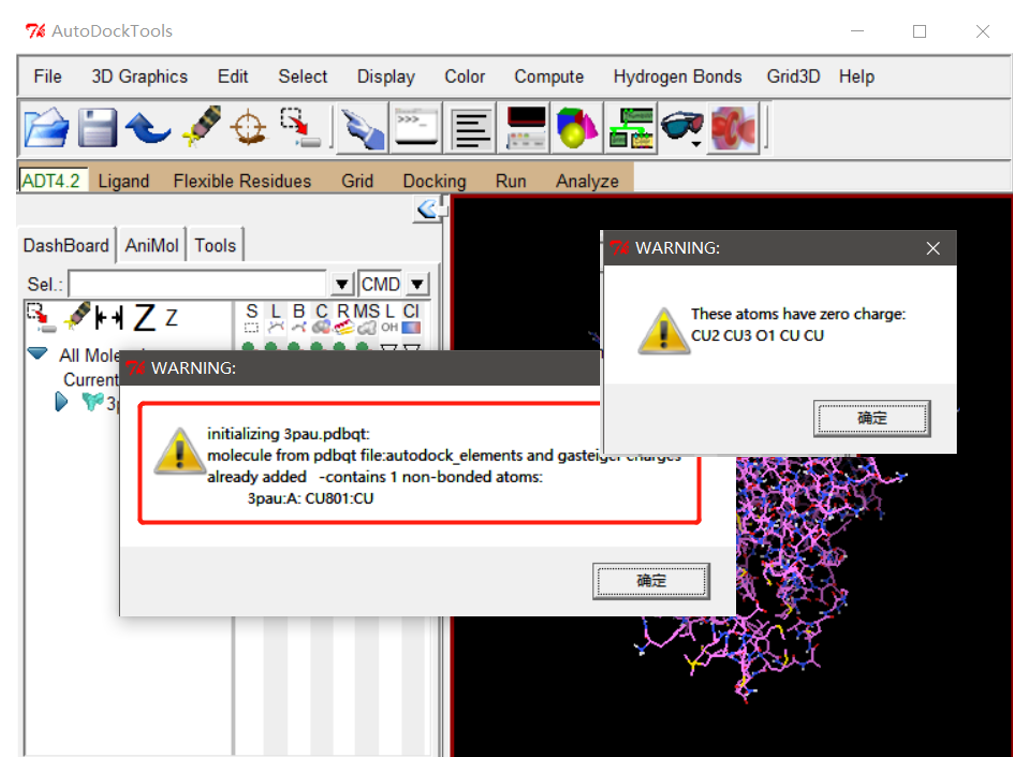
我们继续用前面的配体往下对接。再输出gpf文件的时候,然后运行Auto Grid后,没有产生相应的map文件,而且会运行不会结束,反正我等了好久,就是不会结束。那么我们这么解决呢?
首先,我们可以查看官方文档
http://autodock.scripps.edu/faqs-help/faq/how-do-i-add-new-atom-types-to-autodock-4

还有就是Google,你可以在https://www.researchgate.net/搜索你可能遇到的问题。类似的问题有很多人会遇到的。下面就有一些类似的问题。
https://www.researchgate.net/post/How-to-add-Cobalt-atomic-parameter-in-autodock-42
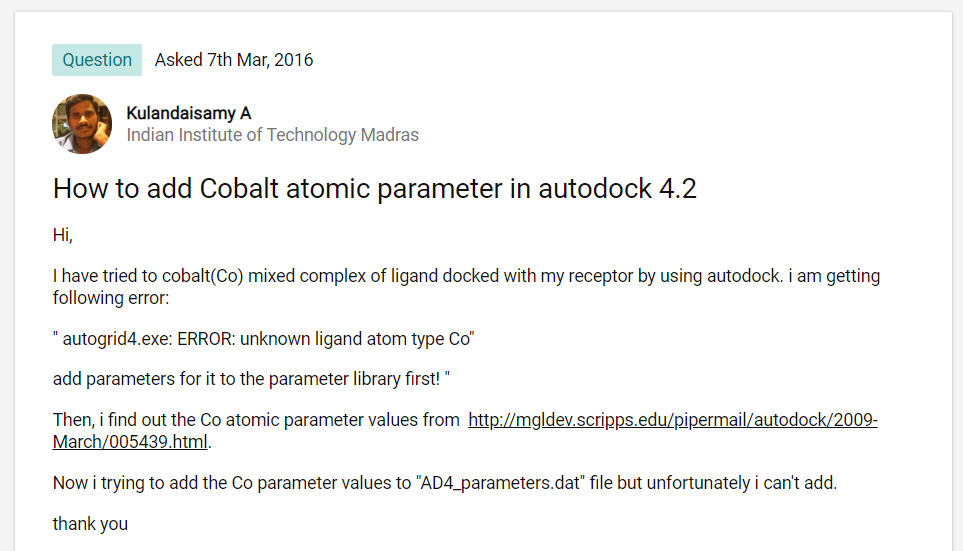
在上面的这个问题中,有一个回答,
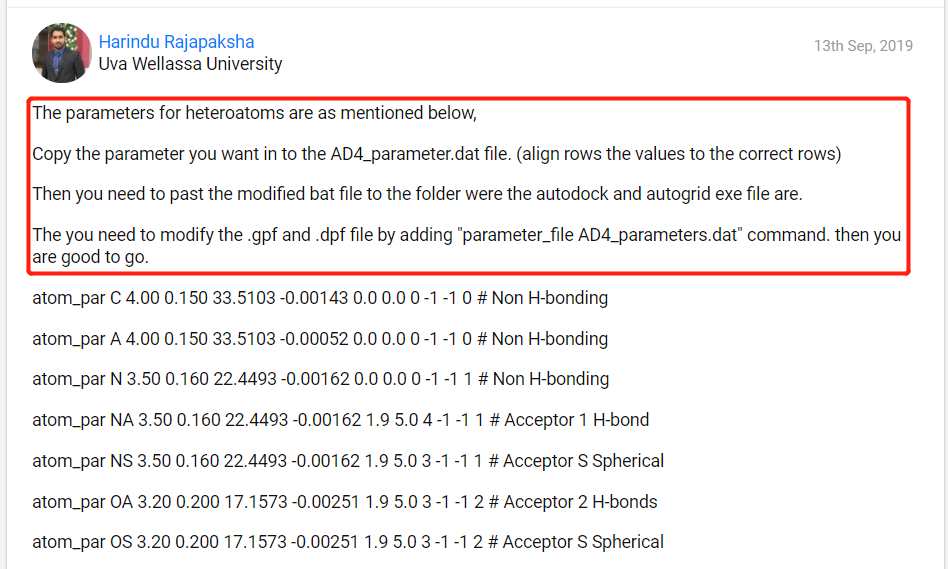
将需要的参数复制到AD4_parameter.dat文件中。(将值对齐到正确的行)
然后你需要把修改过的bat文件放到autodock和autogrid exe文件所在的文件夹中。
然后我们再通过修改.gpf和.dpf文件。再运行。
具体怎么操作,首先,复制AD4_parameter.dat这个文件到工作目录(F:\AutoDock),最好不要在原来的文件下面更改。
atom_par C 4.00 0.150 33.5103 -0.00143 0.0 0.0 0 -1 -1 0 # Non H-bonding
atom_par A 4.00 0.150 33.5103 -0.00052 0.0 0.0 0 -1 -1 0 # Non H-bonding
atom_par N 3.50 0.160 22.4493 -0.00162 0.0 0.0 0 -1 -1 1 # Non H-bonding
atom_par NA 3.50 0.160 22.4493 -0.00162 1.9 5.0 4 -1 -1 1 # Acceptor 1 H-bond
atom_par NS 3.50 0.160 22.4493 -0.00162 1.9 5.0 3 -1 -1 1 # Acceptor S Spherical
atom_par OA 3.20 0.200 17.1573 -0.00251 1.9 5.0 3 -1 -1 2 # Acceptor 2 H-bonds
atom_par OS 3.20 0.200 17.1573 -0.00251 1.9 5.0 3 -1 -1 2 # Acceptor S Spherical
atom_par SA 4.00 0.200 33.5103 -0.00214 2.5 1.0 5 -1 -1 6 # Acceptor 2 H-bonds
atom_par S 4.00 0.200 33.5103 -0.00214 0.0 0.0 0 -1 -1 6 # Non H-bonding
atom_par H 2.00 0.020 0.0000 0.00051 0.0 0.0 0 -1 -1 3 # Non H-bonding
atom_par HD 2.00 0.020 0.0000 0.00051 0.0 0.0 2 -1 -1 3 # Donor 1 H-bond
atom_par HS 2.00 0.020 0.0000 0.00051 0.0 0.0 1 -1 -1 3 # Donor S Spherical
atom_par P 4.20 0.200 38.7924 -0.00110 0.0 0.0 0 -1 -1 5 # Non H-bonding
atom_par Br 4.33 0.389 42.5661 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par BR 4.33 0.389 42.5661 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ca 1.98 0.550 2.7700 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par CA 1.98 0.550 2.7700 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cl 4.09 0.276 35.8235 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-binding
atom_par CL 4.09 0.276 35.8235 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-binding
atom_par F 3.09 0.080 15.4480 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-binding
atom_par Fe 1.30 0.010 1.8400 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par FE 1.30 0.010 1.8400 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par I 4.72 0.550 55.0585 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-binding
atom_par Mg 1.30 0.875 1.5600 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par MG 1.30 0.875 1.5600 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Mn 1.30 0.875 2.1400 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par MN 1.30 0.875 2.1400 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Zn 1.48 0.550 1.7000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par ZN 1.48 0.550 1.7000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par He 2.36 0.056 15.240 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Li 2.45 0.025 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Be 2.76 0.085 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par B 4.08 0.180 12.052 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ne 3.24 0.042 15.440 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Na 3.98 0.030 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Al 4.49 0.505 11.278 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Si 4.30 0.402 12.175 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par K 3.81 0.035 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Sc 3.30 0.019 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ti 3.18 0.017 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par V 3.14 0.016 12.000 -0.00110 0.0 0.0 0 -1 -1 1 # Non H-bonding
atom_par Co 2.87 0.014 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ni 2.83 0.015 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cu 3.50 0.005 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ga 4.38 0.415 11.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ge 4.28 0.379 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par As 4.23 0.309 13.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Se 4.21 0.291 14.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Kr 4.14 0.220 16.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Rb 4.11 0.040 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Sr 3.64 0.235 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Y 3.35 0.072 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Zr 3.12 0.069 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Nb 3.17 0.059 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Mo 3.05 0.056 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Tc 3.00 0.048 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ru 2.96 0.056 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Rh 2.93 0.053 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pd 1.34 0.048 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ag 3.15 0.036 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cd 2.85 0.228 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par In 4.46 0.599 11.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Sn 4.39 0.567 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Sb 4.42 0.449 13.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Te 4.47 0.398 14.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Xe 4.40 0.332 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cs 4.52 0.045 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ba 3.70 0.364 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par La 3.52 0.017 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ce 3.56 0.013 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pr 3.61 0.010 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Nd 3.58 0.010 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pm 3.55 0.009 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Sm 3.52 0.008 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Eu 3.49 0.008 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Gd 3.37 0.009 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Tb 3.45 0.007 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Dy 3.43 0.007 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ho 3.41 0.007 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Er 3.39 0.007 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Tm 3.37 0.006 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Yb 3.36 0.228 12.000 -0.00110 0.0 0.0 0 -1 -1 1 4 # Non H-bonding
atom_par Lu 3.64 0.041 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Hf 3.41 0.072 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ta 3.71 0.081 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par W 3.07 0.067 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Re 2.95 0.066 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Os 3.12 0.120 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ir 2.84 0.073 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pt 2.75 0.080 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Au 3.29 0.039 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Hg 2.71 0.385 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Tl 4.35 0.680 11.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pb 4.30 0.663 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Bi 4.37 0.518 13.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Po 4.71 0.325 14.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par At 4.75 0.284 15.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Rn 4.77 0.248 16.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Fr 4.90 0.050 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ra 3.68 0.404 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Ac 3.48 0.033 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Th 3.40 0.026 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pa 3.42 0.022 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par U 3.40 0.022 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Np 3.42 0.019 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Pu 3.42 0.016 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Am 3.38 0.014 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cm 3.33 0.014 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Bk 3.34 0.013 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Cf 3.31 0.013 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par E 3.30 0.012 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
atom_par Fm 3.29 0.012 12.000 -0.00110 0.0 0.0 0 -1 -1 4 # Non H-bonding
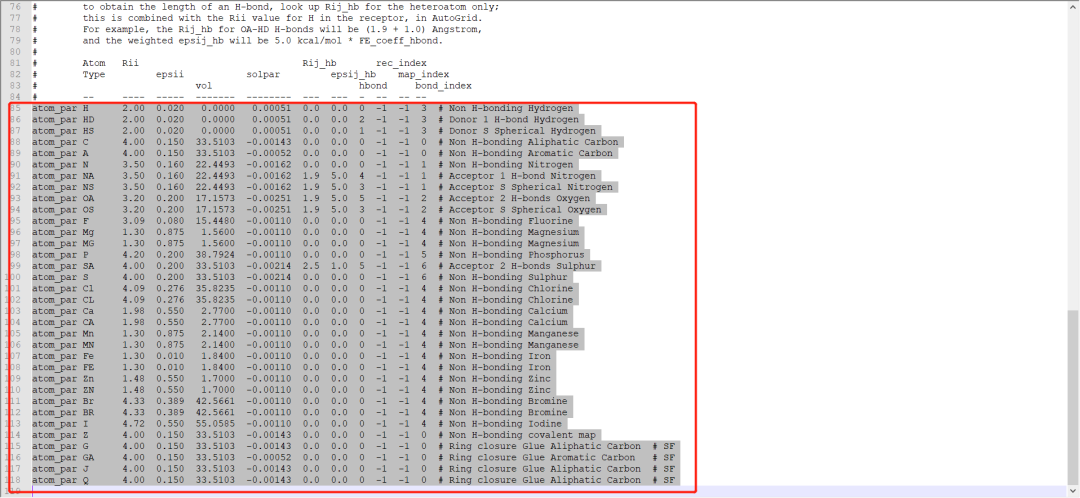
将上面的原子参数复制到AD4_parameter.dat文件中相应的位置。就是下图中红框的区域。然后保存。
我们再生产gpf文件后,先不要往下运行,我们用文本软件打开gpf文件。
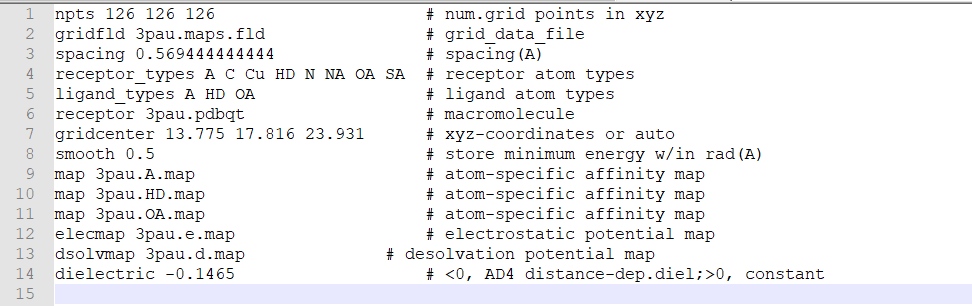
我们在第一行回车后添加一行:parameter_file AD4_parameters.dat。其实就是指定参数文件
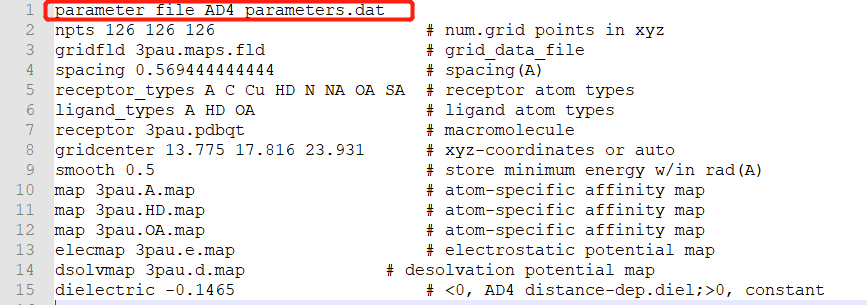
然后我们在运行Auto Grid,接着一直往下,生成dpf文件后,同样用文本软件打开dpf文件,在首行同样添加parameter_file AD4_parameters.dat,保存后再往下运行AutoDock。

这样就不会出错了。当然,实在不想修改文件,麻烦,你可以选择把修改后的文件替换换来的文件,就不需要每次都修改这些文件信息啦。
上面这种处理方式,适合你能找到原子参数的情况下,假设你找不到你要的原子参数,上面这种方法就不可行。但这些原子几乎都够你用啦,放心吧。
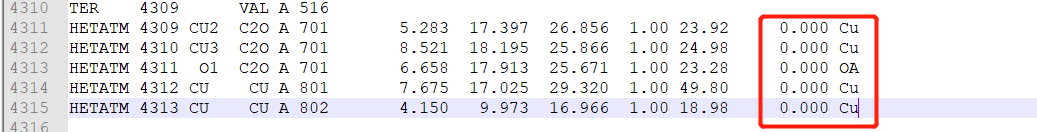
还有一种方法是给金属离子添加形式电荷,我个人觉得有上面方法,就不需要这种,感觉人为添加,加几个?感觉不靠谱,但做法就是打开一开始准备的pdbqt文件。找你离子,你会发现电荷是0,这也是为什么一开始处理会提示电荷为0的原因,这里你可以更改,就是人为的改变。后面就不会出错啦。
好了,关于一下常见问题就介绍到这里,如果你还有错误,那么官方文档是最好的解决办法。
http://autodock.scripps.edu/faqs-help
这里会有各种问题的解决方法。如果对接是你科研的主要内容,那么你需要掌握的不止这些。
参考:
https://www.researchgate.net/post/How-to-add-Cobalt-atomic-parameter-in-autodock-42

这篇关于分子对接教程 | (7) AutoDock对接中易错问题的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!









