本文主要是介绍yll duqiang 制作geo三个独立数据集IPF基因集合 用于分析某个基因是否与生存期相关THBS2 mmp7,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
制作IPF基因集合 用于分析某个基因是否与生存期相关
load("G:/r/duqiang_IPF/surval_analysis_3_independent_dataset_IPF/combined_data_for_surval.RDdata")1#输入想要查询的基因名称或者向量
gene_interested="MMP7" #输入想要查询的基因名称或者向量
library(stringr)
gene_interested=readClipboard() %>% str_split(pattern = ",",gene_interested)[[1]]2#首先查看基因是否存在数据集中,如果不存在则去掉该基因
table( gene_interested %in% rownames(expr.17077clean) & gene_interested %in% rownames(expr.freibrug.IPF))#制作phedata数据用于存活分析
if(1==1){#制作phedata数据用于存活分析for (eachgene in gene_interested) {phe.freigbrug[paste0(eachgene)]=ifelse(expr.freibrug.IPF[eachgene,]>median(expr.freibrug.IPF[eachgene,]),"High",'Low')}head(phe.freigbrug)for (eachgene in gene_interested) {phe.senia[paste0(eachgene)]=ifelse(expr.siena.IPF[eachgene,]>median(expr.siena.IPF[eachgene,]),"High",'Low')}head(phe.senia)for (eachgene in gene_interested) {phe.17077[paste0(eachgene)]=ifelse(expr.17077clean[eachgene,]>median(expr.17077clean[eachgene,]),"High",'Low')}head(phe.17077)}###开始合并三个数据集的phe数据phe_final_3=rbind(phe.freigbrug,phe.senia,phe.17077)
dim(phe_final_3) #[1] 176 5
head(phe_final_3)library(dplyr)
phe_final_3=phe_final_3 %>% transform(time=as.numeric(time))%>% transform(event=as.numeric(event))
getwd()#批量基因差异分析
library(survival)
library(survminer)
for (eachgene in gene_interested) {p=ggsurvplot(survfit(Surv(time, event)~phe_final_3[,eachgene], data=phe_final_3), conf.int=F, pval=TRUE)pdf(paste0(eachgene, "_surval_analysis_from_3_institutes.pdf"),width = 5, height = 5)print(p, newpage = FALSE)dev.off()}
THBS2
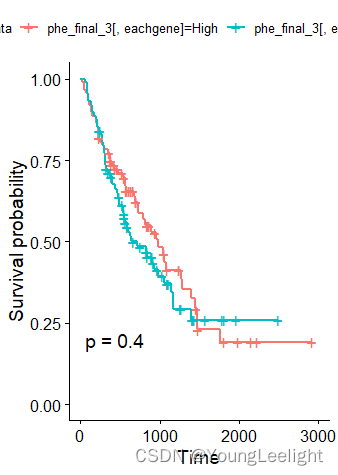
ASB2
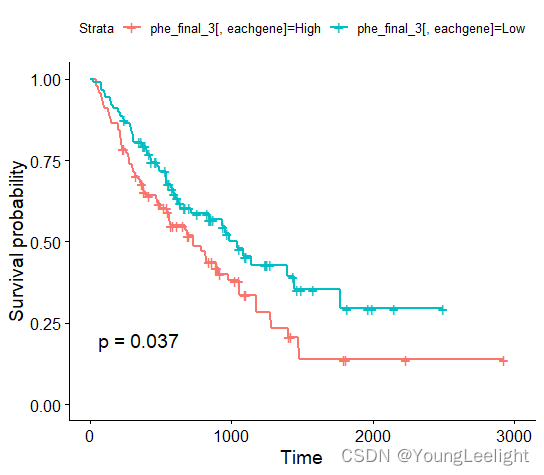
MMP7
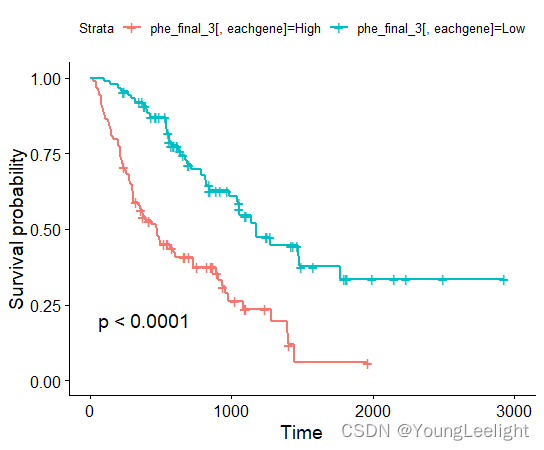
if(1==1){#读取感兴趣的基因gene_interested=readClipboard()head(gene_interested)library(stringr)gene_interested=str_split(pattern = ",",gene_interested)[[1]]gene_interested=gene_interested[-which(gene_interested=="RAB40A")]gene_interested#gpl14550load(file ="G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/Rdatafor_freibrug.RData")head(expr.freiburg_clean)[,1:4]head(meta.14550)[,1:4]dim(expr.freiburg)dim(meta.14550)exprSet.114550.ipf=expr.freiburg[,which(colnames(expr.freiburg)=="GSM1820739"):which(colnames(expr.freiburg)=="GSM1820850")]dim(exprSet.114550.ipf) #[1] 20330 112head(exprSet.114550.ipf)[,1:4]#ID 转换if(1==1){ids14550=data.table::fread("G:/r/duqiang_IPF/GSE70866_BAL_IPF_donors_RNA-seq/GPL14550-9757.txt",)##读取head(ids14550)colnames(ids14550)ids14550=ids14550[,c("ID","GENE","GENE_SYMBOL")]head(ids14550)colnames(ids14550) <- c("PROBE_ID","Entrez_ID", "SYMBOL_ID")#改名,让他适合下面的自定义函数#自建函数p2g <- function(eset,probe2symbol){library(dplyr)library(tibble)library(tidyr)eset <- as.data.frame(eset)p2g_eset <- eset %>% rownames_to_column(var="PROBE_ID") %>% #合并探针的信息inner_join(probe2symbol,by="PROBE_ID") %>% #去掉多余信息select(-PROBE_ID) %>% #重新排列dplyr::select(SYMBOL_ID,everything()) %>% #求出平均数(这边的点号代表上一步产出的数据)mutate(rowMean = rowMeans(.[grep("GSM", names(.))])) %>% #去除symbol中的NAfilter(SYMBOL_ID != "NA") %>% #把表达量的平均值按从大到小排序arrange(desc(rowMean)) %>% # symbol留下第一个distinct(SYMBOL_ID,.keep_all = T) %>% #反向选择去除rowMean这一列dplyr::select(-rowMean) %>% # 列名变成行名column_to_rownames(var = "SYMBOL_ID")#save(p2g_eset, file = "p2g_eset.Rdata")return(p2g_eset)}p2g_eset <- p2g(eset = exprSet.114550.ipf, probe2symbol = ids14550)head(p2g_eset)exprSet.114550.ipf=p2g_eset[,!colnames(p2g_eset)=="Entrez_ID"]}head(exprSet.114550.ipf)[,1:4]colnames(meta.14550)=c('event','time','sex','diagnosis')head(meta.14550)[,1:4]meta.14550=meta.14550[rownames(meta.14550) %in% colnames(exprSet.114550.ipf),]head(meta.14550)[,1:4]dim(meta.14550) #[1] 112 7dim(exprSet.114550.ipf) #[1] 20330 112head(exprSet.114550.ipf)[,1:4]phe.14550=transform(meta.14550,event=as.numeric(event)) %>% transform(time=as.numeric(time)) phe.14550=phe.14550[,1:3]head(phe.14550)exprSet.114550=exprSet.114550.ipf %>% transform(as.numeric()) %>% as.matrix()head(exprSet.114550)[,1:3]for (eachgene in gene_interested) {phe.14550[paste0(eachgene)]=ifelse(exprSet.114550[eachgene,]>median(exprSet.114550[eachgene,]),"High",'Low')}head(phe.14550)dim(phe.14550)dim(phe.17077)##gpl17077load(file ="G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/expr17077.RData")head(expr.17077clean)dim(expr.17077clean) #[1] 20190 64head(meta.17077)colnames(meta.17077)=colnames(meta.14550)head(meta.17077)meta.17077=meta.17077[,1:3]head(meta.17077)head(expr.17077clean)[,1:3]library(dplyr)phe.17077=meta.17077head(meta.17077)exprSet.17077=expr.17077clean %>% as.matrix() %>% transform(as.numeric()) #数据格式转换head(exprSet.17077)[,1:3]for (eachgene in gene_interested) {phe.17077[paste0(eachgene)]=ifelse(exprSet.17077[eachgene,]>median(exprSet.17077[eachgene,]),"High",'Low')}head(phe.17077)##开始合并两个平台的phe数据phe_final_3=rbind(phe.14550,phe.17077)dim(phe_final_3) #[1] 176 37getwd()dir.create("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three")setwd("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three")head(phe_final_3)phe_final_3=phe_final_3 %>% transform(time=as.numeric(time))%>% transform(event=as.numeric(event))getwd()#save(phe_final_3,meta.14550,meta.17077,expr.17077clean,exprSet.114550,file = "G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three/3-institutes.RData")load("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three/3-institutes.RData")#批量基因差异分析for (eachgene in gene_interested) {p=ggsurvplot(survfit(Surv(time, event)~phe_final_3[,eachgene], data=phe_final_3), conf.int=F, pval=TRUE)pdf(paste0(eachgene, "_surval_analysis_from_3_institutes.pdf"),width = 5, height = 5)print(p, newpage = FALSE)dev.off()}load("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three/3-institutes.RData")#批量基因差异分析head(phe_final_3)}load("G:/r/duqiang_IPF/GSE70866—true—_BAL_IPF_donors_RNA-seq/survival_for_genes-three/3-institutes.RData")colnames(exprSet.114550)
nrow(meta.14550)
dim(exprSet.114550) #[1] 20190 112if(1==1){head(meta.14550)expr.freibrug.IPF=exprSet.114550[,which(colnames(exprSet.114550)=="GSM1820739"):which(colnames(exprSet.114550)=="GSM1820800")]meta.freibrug.IPF=meta.14550[1:62,]expr.siena.IPF=exprSet.114550[,!(colnames(exprSet.114550) %in% colnames(expr.freibrug.IPF)) ]meta.siena.IPF=meta.14550[rownames(meta.14550) %in% colnames(expr.siena.IPF),]head(meta.siena.IPF)dim(meta.siena.IPF) #[1] 50 7dim(expr.17077clean) #[1] 20190 64head(meta.17077)colnames(meta.17077)=c("time","event","sex","diagnosis")head(meta.17077)meta.17077=meta.17077[,1:4] %>%select(event,everything())meta.14550=meta.14550[,1:4]head(meta.14550)meta.freibrug.IPF=meta.freibrug.IPF[,1:4]meta.siena.IPF=meta.siena.IPF[,1:4]head(meta.17077)dim(expr.17077clean)# [1] 20190 64identical(rownames(expr.freibrug.IPF),rownames(expr.17077clean))phe.freigbrug=meta.freibrug.IPFphe.senia=meta.siena.IPFphe.17077=meta.17077expr.17077clean=as.matrix(expr.17077clean)getwd()dir.create("G:/r/duqiang_IPF/surval_analysis_3_independent_dataset_IPF")setwd("G:/r/duqiang_IPF/surval_analysis_3_independent_dataset_IPF")save(expr.freibrug.IPF, phe.freigbrug,expr.siena.IPF, phe.senia,expr.17077clean, phe.17077,file ="G:/r/duqiang_IPF/surval_analysis_3_independent_dataset_IPF/combined_data_for_surval.RDdata" )}这篇关于yll duqiang 制作geo三个独立数据集IPF基因集合 用于分析某个基因是否与生存期相关THBS2 mmp7的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






