本文主要是介绍生物系统学中的进化树构建和分析R工具包V.PhyloMaker2的介绍和详细使用,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
V.PhyloMaker2是一个R语言的工具包,专门用于构建和分析生物系统学中的进化树(也称为系统发育树或phylogenetic tree)。以下是对V.PhyloMaker2的一些基本介绍和使用说明:
论文介绍:V.PhyloMaker2: An updated and enlarged R package that can generate very large phylogenies for vascular plants - ScienceDirect
github仓库代码:jinyizju/V.PhyloMaker2: This package (an updated version of 'V.PhyloMaker') can generate a phylogenetic tree for vascular plants based on three different botanical nomenclature systems. (github.com)
介绍:
V.PhyloMaker2提供了一系列的函数和方法,帮助用户处理和分析分子序列数据,包括但不限于:
- 数据预处理:对分子序列数据进行质量控制、格式转换和多重比对。
- 进化树构建:支持多种流行的进化树构建方法,如最大似然法(Maximum Likelihood)、贝叶斯推断法(Bayesian Inference)等。
- 进化树优化:通过搜索最优的树形结构和参数组合来提高进化树的准确性。
- 进化树可视化:提供丰富的图形选项来定制和美化进化树的显示。
- 树形数据分析:包括节点支持度评估、分支长度分析、祖先状态重建等。
详细使用:
由于V.PhyloMaker2的具体使用会涉及到具体的代码操作和数据分析过程,以下是一些基本的使用步骤:
-
安装V.PhyloMaker2: 在R环境中,使用
install.packages("V.PhyloMaker2")命令来安装这个包。#BioManager安装 if (!requireNamespace("BiocManager", quietly = TRUE))install.packages("BiocManager") BiocManager::install("V.PhyloMaker2")#github 安装 install.packages("devtools")library(devtools) install_github("JinYongJiang/V.PhyloMaker") -
加载V.PhyloMaker2: 安装后,使用
library(V.PhyloMaker2)命令来加载这个包。 -
数据预处理: 根据你的数据类型和格式,使用相应的函数进行数据导入和预处理。例如,如果你的数据是fasta格式的序列文件,可以使用
read.FASTA()函数将其读入R。# 导入数据:首先,你需要将你的序列数据导入到R中。这通常是以fasta或 nexus格式存储的。 library(ape) sequences <- read.fasta("your_file.fasta")#数据清理:检查并处理缺失数据、异质性(例如,核苷酸替换)、和错误。 # 查看是否存在任何缺失数据 sum(is.na(sequences))# 如果存在缺失数据,可以考虑删除含有缺失数据的行 sequences <- sequences[!apply(sequences, 1, function(x) any(is.na(x))), ]# 或者用某种方法填补缺失数据(例如,通过平均或中位数) sequences[is.na(sequences)] <- median(sequences, na.rm = TRUE) -
多重比对: 使用
muscle()或其他比对函数对序列进行比对。#序列对齐:对于DNA或蛋白质序列,你需要进行序列对齐。 aligned_sequences <- muscle(sequences)#转换为距离矩阵:将对齐后的序列转换为距离矩阵,这通常是后续构建系统发育树的步骤。 dist_matrix <- dist.dna(aligned_sequences) -
进化树构建: 使用
build.tree()或其他相关函数,根据你的数据和研究目标选择合适的树构建方法。# 假设您已经有了一个包含序列数据的数据框df,并且列名是物种名称 # df <- data.frame(sequence1, sequence2, ..., sequenceN) # 或前面的 data_matrix# 使用build.tree()函数构建进化树 # 这里的参数是假设的,实际参数需要参考V.PhyloMaker包的文档 tree <- build.tree(data = df(或data_matrix), seq_type = "dna", # 数据类型,可以是"dna"、"rna"或"protein"method = "neighbor_joining", # 构建树的方法,例如"neighbor_joining"(邻接法)或"maximum_likelihood"(最大似然法)distance_method = "kimura") # 距离计算方法,例如"kimura"(金氏距离) -
进化树优化: 对构建的初步树进行优化,例如使用
optimize.tree()函数。# 假设你已经使用 build.tree() 建立了一个决策树模型 # 假设 tree_model 是你建立的模型# 查看建立的树的概况 summary(tree_model)# 根据交叉验证选择最佳的剪枝参数 prune_model <- prune.tree(tree_model)# 查看剪枝后的树的概况 summary(prune_model)# 如果需要,你可以根据需要进一步调整剪枝参数 -
进化树可视化: 使用
plot.tree()函数将进化树可视化,并通过调整各种参数来定制图形。# 可视化决策树并调整参数 plot(tree_model, type = "uniform", fsize = 0.8, cex = 0.8, label = "all")# 添加各种参数以定制图形 plot(my_tree,type = "fan", # 树的类型,可以是"phylogram"(分支长度代表进化时间)、"cladogram"(所有分支长度相等)或"fan"(扇形树)show.tip.label = TRUE, # 是否显示叶节点的标签edge.width = 2, # 分支线的宽度edge.color = "black", # 分支线的颜色tip.color = "blue", # 叶节点的颜色no.margin = TRUE, # 是否移除图形边框cex = 0.8, # 标签的字体大小font = 2, # 标签的字体类型main = "My Evolutionary Tree", # 图形的标题sub = "Customized with plot() function") # 图形的副标题 -
树形数据分析: 根据你的研究问题,选择相应的函数进行树形数据分析,如节点支持度评估、分支长度分析等。
# 安装并加载相关包 install.packages("ape") install.packages("phytools") library(ape) library(phytools)# 假设 tree 是你的树形数据# 计算节点支持度 bootstrap_tree <- bootstrap.phylo(tree, FUN = your_function_for_tree, B = 100) # your_function_for_tree 是用于估计树的函数# 生成共识树 consensus_tree <- consensus(bootstrap_tree)# 计算树的相似性矩阵 coph_matrix <- cophenetic(tree)# 绘制共演化历史图 cophyloplot(tree1, tree2)
补充分析示例:
树形数据分析可以使用R中的多个包来实现,例如ape、phangorn、ggtree等。下面是一个简单的示例代码,使用了ape包来进行树形数据分析。
首先,我们需要安装并加载ape包:
install.packages("ape")
library(ape)
接下来,我们可以根据需求读取树形数据。假设我们有一棵简单的进化树,包含5个物种,并且我们想要计算节点的支持度值:
# 创建一个简单的进化树
tree <- rtree(5)# 计算节点的支持度值
supports <- node.depths(tree)
接下来,我们可以绘制树形图,并标记节点的支持度值:
# 绘制树形图
plot(tree, show.node.label = TRUE)# 标记节点支持度值
nodelabels(round(supports, 2), bg = "white")
要分析分支长度,我们可以使用cophenetic.phylo()函数计算树的协同形态矩阵,然后使用plot()函数绘制分支长度图:
# 计算协同形态矩阵
cophenetic_matrix <- cophenetic(tree)# 绘制分支长度图
plot(cophenetic_matrix, main = "Branch Lengths", xlab = "Pairwise Distances")
相似工具包S.PhyloMaker
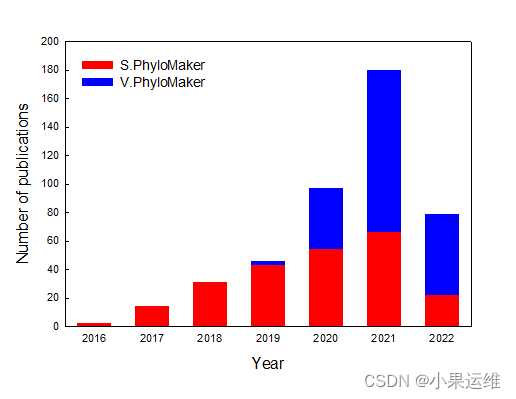
V.PhyloMaker是一个R语言包,由Jin Y和Qian H在2019年开发,主要用于生成大型的植物系统发育树(phylogenies)。这个包特别适用于血管植物(vascular plants)的大规模数据集分析。以下是对V.PhyloMaker的介绍:
V.PhyloMaker的主要功能包括:
-
大规模数据处理:该软件包能够处理大规模的基因序列或单核苷酸多态性(SNP)数据,这对于构建包含大量物种的系统发育树是非常重要的。
-
系统发育树构建:V.PhyloMaker采用多种算法和方法来推断物种之间的进化关系,这些方法可能包括最大似然法、贝叶斯推理法以及其他基于距离的方法。
-
模型选择:该软件包允许用户根据他们的数据特性选择最适合的序列进化模型,这有助于提高构建的系统发育树的准确性。
-
可视化和分析:虽然V.PhyloMaker本身可能不直接提供高级的可视化功能,但因为它是在R环境中运行的,所以可以轻松地与R中的其他图形和可视化包(如ggplot2或ape)结合使用,以生成和注释系统发育树。
-
数据整合:V.PhyloMaker能够导入和导出各种格式的数据,使得它与其他生物信息学工具和平台具有良好的兼容性。
这篇关于生物系统学中的进化树构建和分析R工具包V.PhyloMaker2的介绍和详细使用的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







