本文主要是介绍PDB Database - 高质量 RCSB PDB 蛋白质结构筛选与过滤,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/132307119
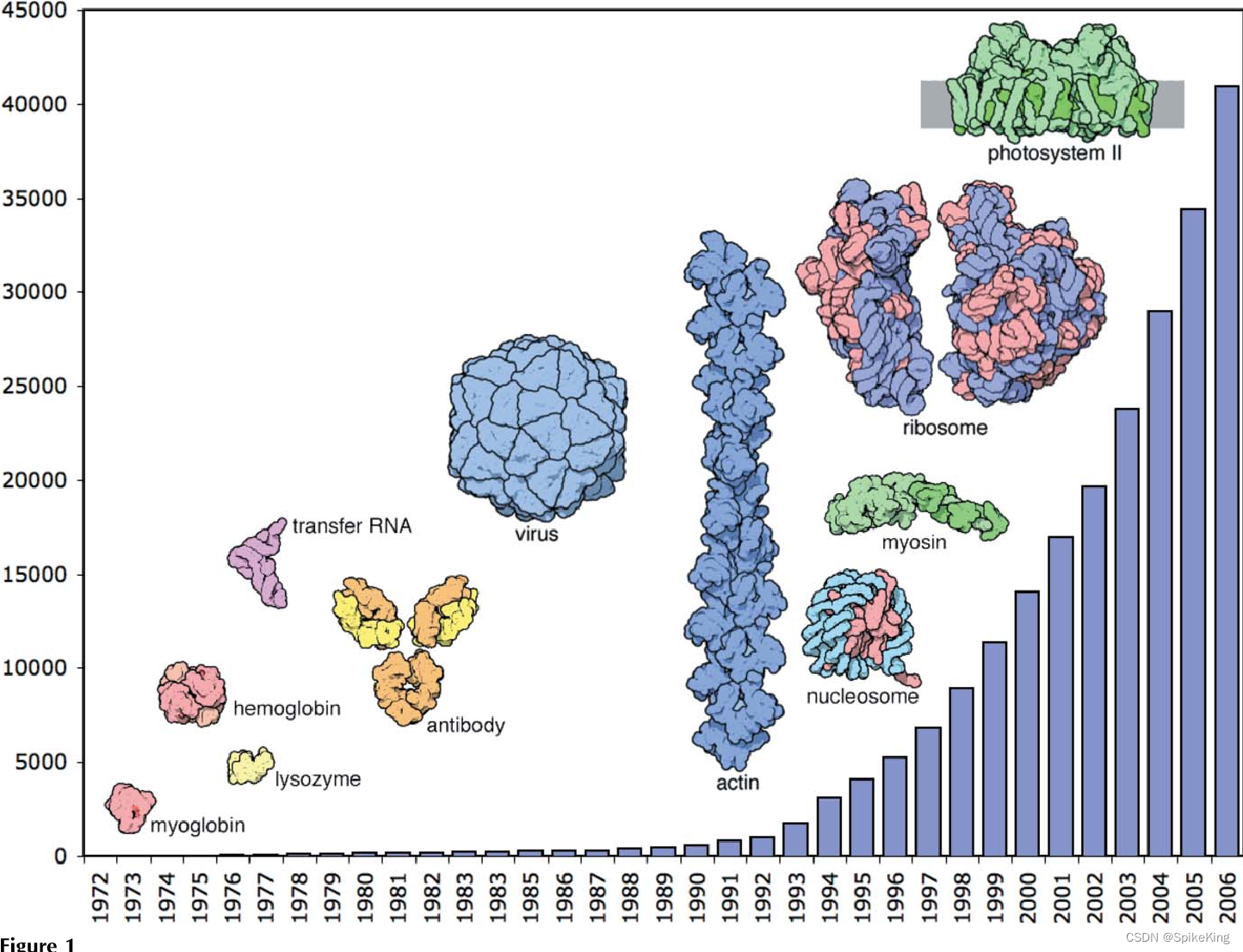
Protein Data Bank (PDB) 是一个收集和存储三维结构数据的公共数据库,主要包括蛋白质和核酸分子。PDB 由美国、欧洲和日本三个机构共同管理,每周更新一次。PDB 的目的是为生物学、生物化学、生物物理学和医学等领域的研究者提供结构信息,促进科学发现和教育。PDB 的数据可以通过网站、FTP 服务器或应用程序接口 (API) 免费获取,也可以通过各种工具和服务进行可视化、分析和下载。
根据不同维度,从 RCSB PDB 筛选与过滤出高质量的数据,用于下游任务的分析与处理,主要包括以下维度:
- 按 发布时间 (Release Date) 过滤。
- 按 单体 (Monomer) 或 多聚体 (Multimer) 过滤。
- 按 氨基酸类型,是RNA\DNA,还是蛋白质 (Protein) 过滤。
- 按 结构分辨率 (Resolution) 过滤。
- 按 相同氨基酸占比 过滤。
- 按 最短蛋白质链长 (Seq. Len.) 过滤。
- 按 实验方法 (Experiment Method) 过滤。
不同的算法,也有不同的过滤规则,具体可以参考 AlphaFold、ESMFold、UniFold 等相关论文。例如 PolyFold:

PDB 数据库的信息,参考:RCSB PDB 数据集 (2023.8) 的多维度信息统计
以全量 PDB 为例,共有 203657 条数据,即:
- 时间
[2021-09-30, 2023-01-01),剩余 15961,过滤 92.1628%。 - 过滤单链,剩余 11621,过滤 27.1913%。
- 过滤非蛋白,剩余 10416,过滤 10.3692%。
- 过滤分辨率,小于 9A,剩余 10325,过滤 0.8737%。
- 过滤重复残基,重复率大于等于0.8,剩余 10063,过滤 2.5375%。
- 过滤序列长度小于20,剩余 9096,过滤 9.6095%。
- 过滤实验方法,保留 XD\SN\EM\EC,剩余 9095,过滤 0.011%。
最终,从多聚体 10416 下降至 9095,保留率 87.32%。
处理脚本命令:
python3 scripts/dataset_generator.py -i data/pdb_base_info_202308.csv -o mydata/dataset/train.csv -b 2021-09-30 -e 2023-01-01
运行日志:
[Info] sample: 203657
[Info] filter [2021-09-30 ~ 2023-01-01): 15961/203657, 92.1628 %
[Info] filter monomer: 11621/15961, 27.1913%
[Info] filter na: 10416/11621, 10.3692%
[Info] filter resolution < 9: 10325/10416, 0.8737%
[Info] filter aa same >= 0.8: 10063/10325, 2.5375%
[Info] filter seq len < 20: 9096/10063, 9.6095%
[Info] filter experiment method (include XD|SN|EM|EC): 9095/9096, 0.011%
源码参考:
#!/usr/bin/env python
# -- coding: utf-8 --
"""
Copyright (c) 2022. All rights reserved.
Created by C. L. Wang on 2023/8/15
"""
import argparse
import ast
import collections
import os
import sys
from pathlib import Pathimport pandas as pdp = os.path.dirname(os.path.dirname(os.path.abspath(__file__)))
if p not in sys.path:sys.path.append(p)from myutils.project_utils import sort_dict_by_value
from root_dir import ROOT_DIR, DATA_DIRclass DatasetGenerator(object):"""根据多个条件,筛选构建 PDB 蛋白质结构的数据集"""def __init__(self):pass@staticmethoddef filter_release_date(df, date=("2021-09-30", "2023-01-01")):"""筛选发布日期"""df = df.sort_values(by=['release_date'])n0 = len(df)print(f"[Info] sample: {len(df)}")n_df = df.loc[(df['release_date'] >= f"{date[0]}") & (df['release_date'] < f"{date[1]}")]n1 = len(n_df)print(f"[Info] filter [{date[0]} ~ {date[1]}): {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)} %")return n_df@staticmethoddef filter_monomer(df):"""筛选过滤 Monomer,保留 Multimer"""n0 = len(df)df_chain_type = df["chain_type"]flags = []for ct_str in df_chain_type:items = ct_str.split(",")if len(items) == 1: # 过滤单链flags.append(False)else:flags.append(True)n_df = df.loc[flags]n1 = len(n_df)print(f"[Info] filter monomer: {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_df@staticmethoddef filter_na(df):"""过滤 RNA、DNA,只保留 Protein"""n0 = len(df)df_chain_type = df["chain_type"]flags = []for ct_str in df_chain_type:items = ct_str.split(",")assert len(items) != 1if len(set(items)) != 1: # 过滤NAflags.append(False)else:flags.append(True)n_df = df.loc[flags]n1 = len(n_df)print(f"[Info] filter na: {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_df@staticmethoddef filter_resolution(df, r_val=9):"""过滤低分辨率"""n0 = len(df)n_df = df.loc[df["resolution"] < r_val]n1 = len(n_df)print(f"[Info] filter resolution < 9: {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_df@staticmethoddef filter_same_aa(df, thr_ratio=0.8):"""过滤相同氨基酸占比较高"""n0 = len(df)df_seq = df["seq"]flags = []for item_str in df_seq:if not isinstance(item_str, str):flags.append(False) # 去掉continueis_same = Falseitems = item_str.split(",")for item in items:n_item = len(item)num_aa_dict = collections.defaultdict(int)for aa in item:num_aa_dict[aa] += 1num_aa_data = sort_dict_by_value(num_aa_dict, reverse=True)v_max = num_aa_data[0][1]ratio = v_max / n_itemif ratio >= thr_ratio:# print(f"[Info] item: {item}")is_same = Truebreakif is_same:flags.append(False) # 去掉else:flags.append(True)n_df = df.loc[flags]n1 = len(n_df)print(f"[Info] filter aa same >= 0.8: {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_df@staticmethoddef filter_short_seq_len(df, thr_len=20):"""过滤序列长度角度的蛋白质,Multimer中只要存在1条,即过滤"""n0 = len(df)df_seq = df["seq"]flags = []for item_str in df_seq:is_short = Falseitems = item_str.split(",")for item in items:if len(item) < thr_len:is_short = Truebreakif is_short:flags.append(False)else:flags.append(True)n_df = df.loc[flags]n1 = len(n_df)print(f"[Info] filter seq len < 20: {n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_df@staticmethoddef filter_experiment_method(df, method_list=("XD", "SN", "EM", "EC")):"""筛选实验条件,目前保留 ["XD", "SN", "EM", "EC"]"""n0 = len(df)experiment_method = df["experiment_method"]flags = []for ex_item in experiment_method:items = ex_item.split(";")is_save = Falsefor item in items:method = item.strip()sub_names = method.split(" ")sub_m = "".join([i[0].upper() for i in sub_names])if sub_m in method_list:is_save = Truebreakif is_save:flags.append(True)else:flags.append(False)n_df = df.loc[flags]n1 = len(n_df)print(f"[Info] filter experiment method (include XD|SN|EM|EC): "f"{n1}/{n0}, {round(100 - (n1/n0 * 100), 4)}%")return n_dfdef filer_pipeline(self, df, date):"""过滤的全部流程"""# 1. 时间筛选df = self.filter_release_date(df, date=date)# 2. 过滤单链df = self.filter_monomer(df)# 3. 过滤非蛋白df = self.filter_na(df)# 4. 分辨率 < 9Adf = self.filter_resolution(df)# 5. 单个氨基酸占比小于 80%df = self.filter_same_aa(df)# 6. 过滤链长 < 20df = self.filter_short_seq_len(df)# 7. 实验方法包括 X-ray Diffraction、Electron Microscopy、Solution NMR、Solid-state NMR、Electron Crystallographydf = self.filter_experiment_method(df)return dfdef process(self, csv_path, output_file, date_range):"""处理数据集"""assert os.path.isfile(csv_path) and output_file.endswith("csv")df = pd.read_csv(csv_path)df.info()# 复制列df = df[["pdb_id", "chain_id", "resolution", "release_date", "seq", "len", "mol", "experiment_method"]].copy()df.columns = ["pdb_id", "chain_id", "resolution", "release_date", "seq", "len","chain_type", "experiment_method"]# 对齐 chain_type 数据格式def func(x):if not isinstance(x, str):return "none"ct_item = ast.literal_eval(x)c_type_list = []for item in ct_item:c_type = item.strip()c_type_list.append(c_type)return ",".join(c_type_list)df["chain_type"] = df["chain_type"].apply(lambda x: func(x))new_df = self.filer_pipeline(df, date=date_range) # 过滤流new_df = new_df.drop('experiment_method', axis=1) # 去掉new_df.to_csv(output_file, index=False)# train_df = self.filer_pipeline(df, date=("2021-09-30", "2023-01-01")) # 过滤流# train_df = train_df.drop('experiment_method', axis=1)# train_df.to_csv(os.path.join(output_dir, "train.csv"), index=False)# val_df = self.filer_pipeline(df, date=("2023-01-01", "2025-01-01")) # 过滤流# val_df = val_df.drop('experiment_method', axis=1)# val_df.to_csv(os.path.join(output_dir, "val.csv"), index=False)print("[Info] over!")def main():parser = argparse.ArgumentParser()parser.add_argument("-i","--input-file",help="the input file of pdb database profile.",type=Path,required=True,)parser.add_argument("-o","--output-file",help="the output file of result csv.",type=Path,required=True)parser.add_argument("-b","--begin-date",help="the begin date of pdb db, i.e. 2021-09-30 .",type=str,required=True)parser.add_argument("-e","--end-date",help="the end date of pdb db, i.e. 2023-01-01 , default 2999-12-31 .",type=str,default="2999-12-31")args = parser.parse_args()input_file = str(args.input_file)output_file = str(args.output_file)begin_date = str(args.begin_date)end_date = str(args.end_date)date_range = (begin_date, end_date)assert os.path.isfile(input_file)dg = DatasetGenerator()# input_file = os.path.join(ROOT_DIR, "data", "pdb_base_info_202308.csv")# output_file = os.path.join(DATA_DIR, "dataset", "train.csv")# date_range = ("2021-09-30", "2023-01-01")# date_range = ("2023-01-01", "2025-01-01") # valdg.process(input_file, output_file, date_range)if __name__ == '__main__':main()
参考
- StackOverflow - Filtering Pandas DataFrames on dates
- Ways to filter Pandas DataFrame by column values
- pandas.DataFrame.sort_values
- StackOverflow - Renaming column names in Pandas
- Pandas: How to Create New DataFrame from Existing DataFrame
- Export Pandas to CSV without Index & Header
- StackOverflow - Delete a column from a Pandas DataFrame
这篇关于PDB Database - 高质量 RCSB PDB 蛋白质结构筛选与过滤的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!








