本文主要是介绍Seurat 源码学习之VlnPlot,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
今天很好奇Seurat里的Vlnplot是怎么画的,花了一个上午研究一下这个画图,其实还是很简单的哈, 以官网的pbmc3k为例
#
library(dplyr)
library(Seurat)
library(patchwork)
setwd("/Users/xiaokangyu/Desktop/Seurat_raw_code/")
pbmc.data = Read10X("/Users/xiaokangyu/Desktop/test_dataset/pbmc_3k/filtered_gene_bc_matrices/hg19/")pbmc = CreateSeuratObject(counts = pbmc.data,project = "pbmc3k",min.cells=3,min.features = 200)
pbmcpbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)pbmc <- NormalizeData(pbmc)pbmc <- FindVariableFeatures(pbmc, selection.method = "vst", nfeatures = 2000)all.genes <- rownames(pbmc)pbmc <- ScaleData(pbmc, features = all.genes)pbmc <- RunPCA(pbmc, features = VariableFeatures(object = pbmc))pbmc <- FindNeighbors(pbmc, dims = 1:10)pbmc <- FindClusters(pbmc, resolution = 0.5)pbmc <- RunUMAP(pbmc, dims = 1:10)new.cluster.ids <- c("Naive CD4 T", "Memory CD4 T", "CD14+ Mono", "B", "CD8 T", "FCGR3A+ Mono", "NK", "DC", "Platelet")names(new.cluster.ids) <- levels(pbmc)pbmc <- RenameIdents(pbmc, new.cluster.ids)pbmc[['cell_type']] <- pbmc@active.identlevels(pbmc)saveRDS(pbmc, file = "./pbmc.rds")# p =DimPlot(pbmc, reduction = "umap", group.by = "cell_type")#画batch图
# print(p)
#
# pp=DimPlot(pbmc, reduction = "umap", group.by = "cell_type", label.size = 5,label=T)+ggtitle("annotated Celltype")+NoLegend()
# print(pp)
#
# print(p+pp)
# p1=FeaturePlot(pbmc, features = c("MS4A1", "GNLY", "CD3E", "CD14", "FCER1A", "FCGR3A", "LYZ", "PPBP",
# "CD8A"))
#
# p2=FeaturePlot(pbmc, features = c("MS4A1", "CD79A"))
# print(p2)
#
# p3=VlnPlot(pbmc, features = c("MS4A1", "CD79A"))p4 = VlnPlot(pbmc,features = c("MS4A1"))print(p4)
# pbmc@active.assay
结果如下
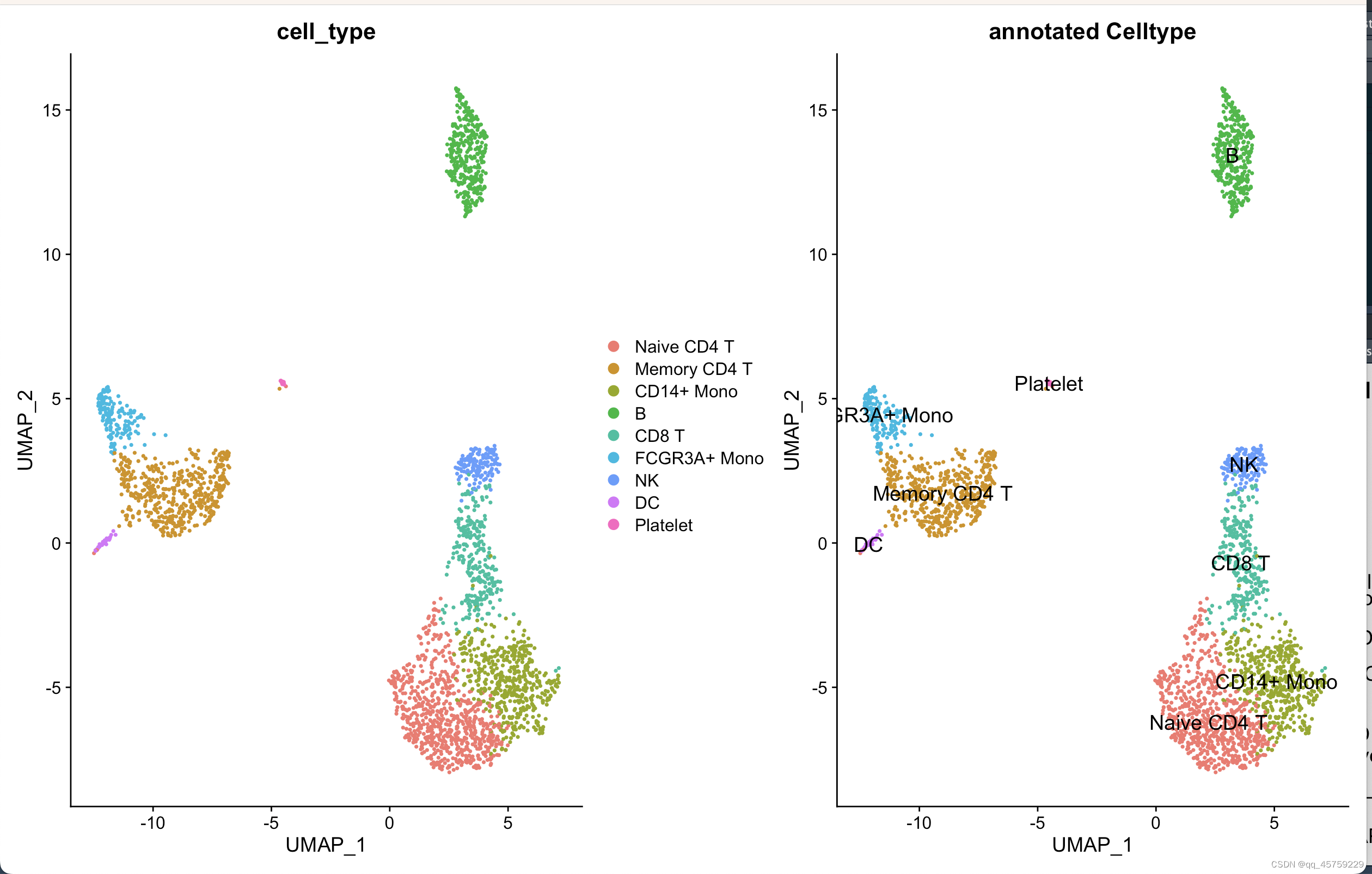
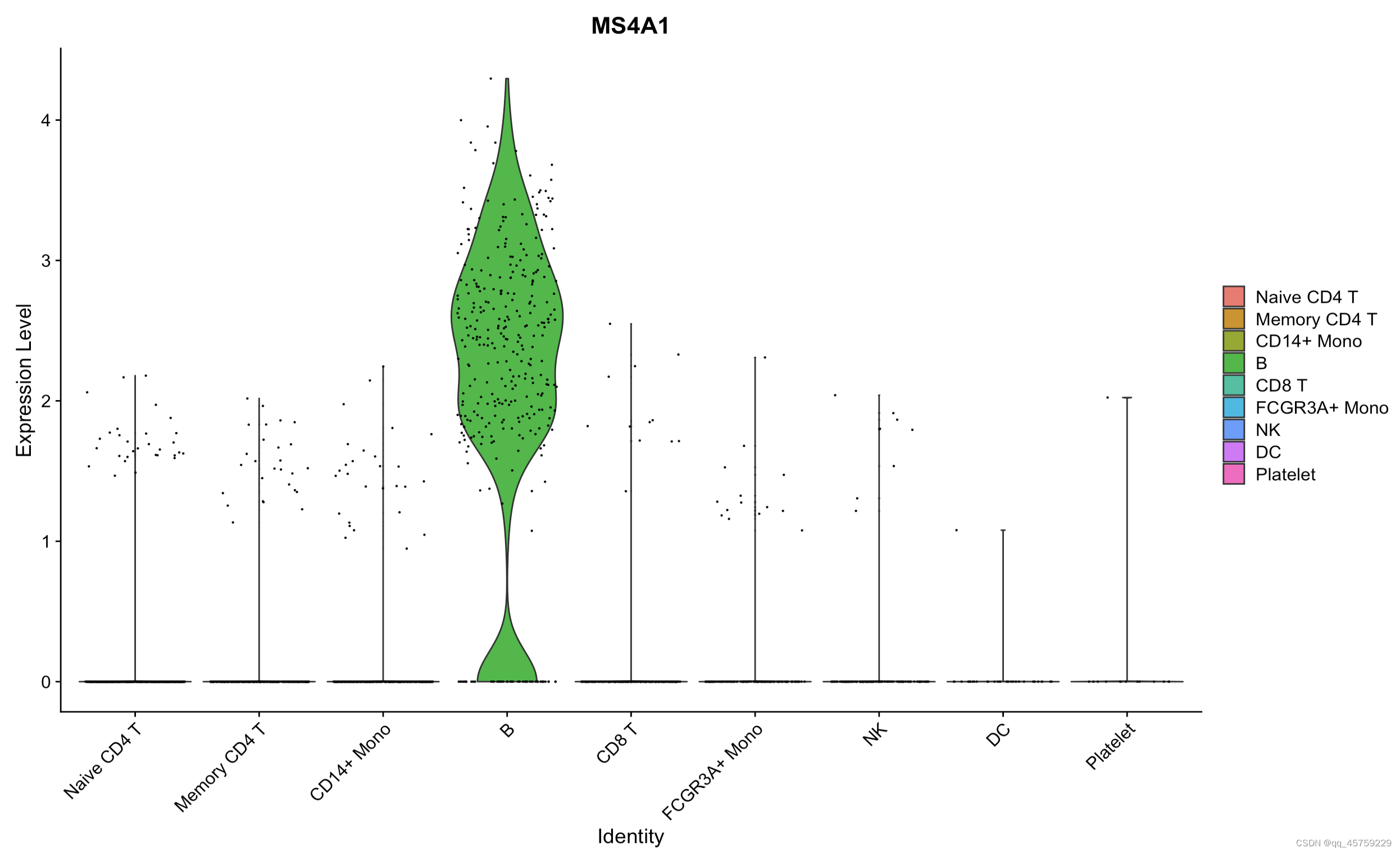
可以看到这个结果很好哈,现在研究这个小提琴图是怎么画的
首先注意一点本地mac电脑可以直接调试,而服务器的rstudio打上断点后没有调试的相关按钮,很奇怪
 而在服务器上
而在服务器上
注意在R里的调试有一个好处,虽然我直接安装了Seurat,直接使用调试按钮直接进入函数,但是python里有的包你是进不去的,首先进入的
 可以看到Vlnplot调用的是ExIplot函数,接着调用的是SingleExIPlot函数
可以看到Vlnplot调用的是ExIplot函数,接着调用的是SingleExIPlot函数
 这里可以看到真正的画图函数,代码还是很简单的
这里可以看到真正的画图函数,代码还是很简单的
下面是我自己复现的代码
rm(list=ls())
library(Seurat)
library(ggplot2)
library(cowplot)pbmc = readRDS("./pbmc.rds")
set.seed(42)
feature ="MS4A1"
data_all = pbmc@assays$RNA@data # 取出来上dgcMatrix是稀疏矩阵,没法取行列
data_df = data.frame(data_all)
data = data.frame(t(data_df[feature,])) # 取feature 基因对应的表达
data$ident =pbmc@meta.data$cell_type # 我这里发现一个R的bug,就是对于因子类型的变量
#如果写入csv文件再读取,那么这个factor对应的level的顺序可能会自己变化,不再是开始保存时的结果了,
#如果要保存level对应的信息时,还是应该使用saveRDS而不是write.csvnoise <- rnorm(n = length(x = data[, feature]))/1e+05
data[, feature] <- data[, feature] + noisept.size = 0.1154295
x="ident"
y="`MS4A1`"
fill ="ident"
xlab="Identity"
feature = "MS4A1"
ylab ="Expression Level"jitter <- geom_jitter(height = 0, size = pt.size, show.legend = FALSE)
plot <- ggplot(data = data, mapping = aes_string(x = x, y = y, fill = fill)[c(2, 3, 1)]) + labs(x = xlab, y = ylab, title = feature, fill = NULL) + theme_cowplot() + theme(plot.title = element_text(hjust = 0.5))geom <- list(geom_violin(scale = "width", adjust = 1, trim = TRUE), theme(axis.text.x = element_text(angle = 45, hjust = 1)))plot <- do.call(what = "+", args = list(plot, geom))plot = plot + jitter
print(plot)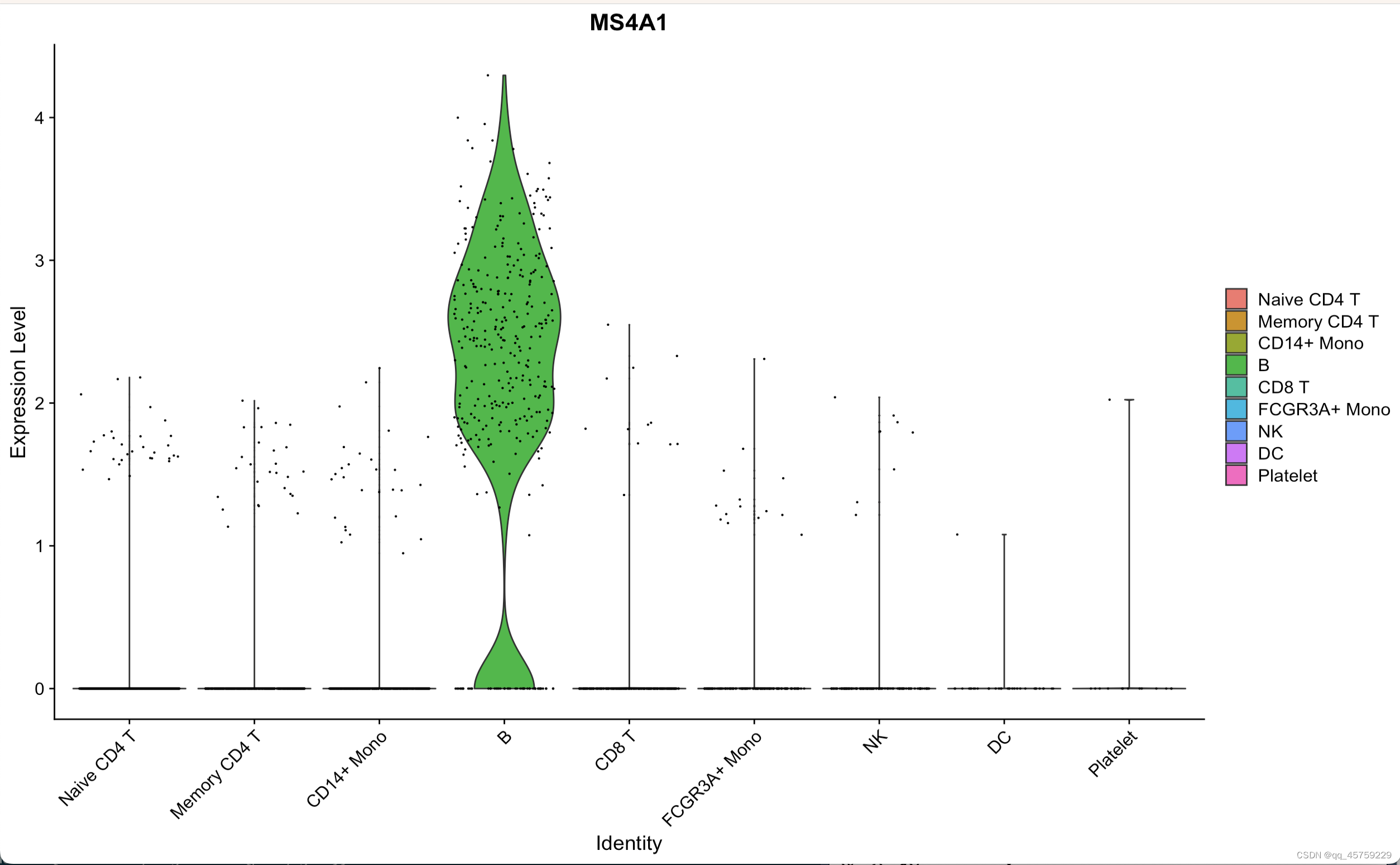 这个结果和Seurat画出来的是一样的,嘿嘿
这个结果和Seurat画出来的是一样的,嘿嘿
我的环境如下
sessionInfo()
R version 4.1.2 (2021-11-01)
Platform: x86_64-apple-darwin17.0 (64-bit)
Running under: macOS 13.1Matrix products: default
LAPACK: /Library/Frameworks/R.framework/Versions/4.1/Resources/lib/libRlapack.dyliblocale:
[1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8attached base packages:
[1] stats4 stats graphics grDevices utils datasets methods base other attached packages:[1] cowplot_1.1.1 patchwork_1.1.2 SeuratObject_4.1.3 [4] Seurat_4.3.0 dplyr_1.1.0 ggplot2_3.4.1 [7] splatter_1.18.2 SingleCellExperiment_1.16.0 SummarizedExperiment_1.24.0
[10] Biobase_2.54.0 GenomicRanges_1.46.1 GenomeInfoDb_1.30.1
[13] IRanges_2.28.0 S4Vectors_0.32.4 BiocGenerics_0.40.0
[16] MatrixGenerics_1.6.0 matrixStats_0.63.0 loaded via a namespace (and not attached):[1] ggbeeswarm_0.7.1 Rtsne_0.16 colorspace_2.0-3 [4] deldir_1.0-6 ellipsis_0.3.2 ggridges_0.5.4 [7] XVector_0.34.0 rstudioapi_0.14 spatstat.data_3.0-0 [10] farver_2.1.1 leiden_0.4.3 listenv_0.9.0 [13] ggrepel_0.9.2 fansi_1.0.3 R.methodsS3_1.8.2 [16] codetools_0.2-18 splines_4.1.2 polyclip_1.10-4 [19] jsonlite_1.8.4 ica_1.0-3 cluster_2.1.2 [22] R.oo_1.25.0 png_0.1-8 uwot_0.1.14 [25] shiny_1.7.4 sctransform_0.3.5 spatstat.sparse_3.0-0 [28] compiler_4.1.2 httr_1.4.4 backports_1.4.1 [31] Matrix_1.5-1 fastmap_1.1.0 lazyeval_0.2.2 [34] cli_3.6.0 later_1.3.0 htmltools_0.5.4 [37] tools_4.1.2 igraph_1.3.5 gtable_0.3.1 [40] glue_1.6.2 GenomeInfoDbData_1.2.7 RANN_2.6.1 [43] reshape2_1.4.4 Rcpp_1.0.10 scattermore_0.8 [46] vctrs_0.5.2 spatstat.explore_3.0-5 nlme_3.1-153 [49] progressr_0.13.0 lmtest_0.9-40 spatstat.random_3.0-1 [52] stringr_1.5.0 globals_0.16.2 mime_0.12 [55] miniUI_0.1.1.1 lifecycle_1.0.3 irlba_2.3.5.1 [58] goftest_1.2-3 future_1.30.0 zlibbioc_1.40.0 [61] MASS_7.3-54 zoo_1.8-11 scales_1.2.1 [64] promises_1.2.0.1 spatstat.utils_3.0-1 parallel_4.1.2 [67] RColorBrewer_1.1-3 reticulate_1.27 pbapply_1.6-0 [70] gridExtra_2.3 ggrastr_1.0.1 stringi_1.7.8 [73] checkmate_2.1.0 BiocParallel_1.28.3 rlang_1.1.0 [76] pkgconfig_2.0.3 bitops_1.0-7 lattice_0.20-45 [79] ROCR_1.0-11 purrr_1.0.1 tensor_1.5 [82] labeling_0.4.2 htmlwidgets_1.6.1 tidyselect_1.2.0 [85] parallelly_1.33.0 RcppAnnoy_0.0.20 plyr_1.8.8 [88] magrittr_2.0.3 R6_2.5.1 generics_0.1.3 [91] DelayedArray_0.20.0 DBI_1.1.3 withr_2.5.0 [94] pillar_1.8.1 fitdistrplus_1.1-8 survival_3.2-13 [97] abind_1.4-5 RCurl_1.98-1.9 sp_1.5-1
[100] tibble_3.2.1 future.apply_1.10.0 crayon_1.5.2
[103] KernSmooth_2.23-20 utf8_1.2.2 spatstat.geom_3.0-3
[106] plotly_4.10.1 locfit_1.5-9.7 grid_4.1.2
[109] data.table_1.14.6 digest_0.6.31 xtable_1.8-4
[112] tidyr_1.3.0 httpuv_1.6.7 R.utils_2.12.2
[115] munsell_0.5.0 beeswarm_0.4.0 viridisLite_0.4.1
[118] vipor_0.4.5
这篇关于Seurat 源码学习之VlnPlot的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






